Análisis de regresión lineal múltiple
Gonzalo Pérez
13 de abril de 2020
3.
El objetivo de los siguientes datos es analizar si el consumo de ciertos alimentos y otros factores (como edad, sexo, etc.) tienen una relación con el nivel de plasma beta-carotene. El interés original de estos datos fue recabar información que pudiera servir para encontrar los factores asociados con niveles bajos de plasma beta-carotene dado que estos podrían estar asociados con el riesgo de desarrollar algunos canceres.
Variables:
- Dependiente. BETAPLASMA, Plasma beta-carotene (ng/ml)
- Explicativas:
- AGE (years)
- SEX (1=Male, 2=Female)
- VITUSE, Vitamin Use (1=Yes, fairly often, 2=Yes, not often, 3=No)
- CALORIES, Number of calories consumed per day.
- FAT, Grams of fat consumed per day.
- FIBER, Grams of fiber consumed per day.
- ALCOHOL, Number of alcoholic drinks consumed per week.
- CHOLESTEROL, Cholesterol consumed (mg per day).
- BETADIET, Dietary beta-carotene consumed (mcg per day).
Datos=read.table("images/archivos/prdata.dat",
header=FALSE, sep="\t")
names(Datos)=c("AGE", "SEX", "SMOKSTAT", "QUETELET", "VITUSE", "CALORIES", "FAT", "FIBER", "ALCOHOL", "CHOLESTEROL", "BETADIET", "RETDIET", "BETAPLASMA", "RETPLASMA")
head(Datos)str(Datos)## 'data.frame': 315 obs. of 14 variables:
## $ AGE : int 64 76 38 40 72 40 65 58 35 55 ...
## $ SEX : int 2 2 2 2 2 2 2 2 2 2 ...
## $ SMOKSTAT : int 2 1 2 2 1 2 1 1 1 2 ...
## $ QUETELET : num 21.5 23.9 20 25.1 21 ...
## $ VITUSE : int 1 1 2 3 1 3 2 1 3 3 ...
## $ CALORIES : num 1299 1032 2372 2450 1952 ...
## $ FAT : num 57 50.1 83.6 97.5 82.6 56 52 63.4 57.8 39.6 ...
## $ FIBER : num 6.3 15.8 19.1 26.5 16.2 9.6 28.7 10.9 20.3 15.5 ...
## $ ALCOHOL : num 0 0 14.1 0.5 0 1.3 0 0 0.6 0 ...
## $ CHOLESTEROL: num 170.3 75.8 257.9 332.6 170.8 ...
## $ BETADIET : int 1945 2653 6321 1061 2863 1729 5371 823 2895 3307 ...
## $ RETDIET : int 890 451 660 864 1209 1439 802 2571 944 493 ...
## $ BETAPLASMA : int 200 124 328 153 92 148 258 64 218 81 ...
## $ RETPLASMA : int 915 727 721 615 799 654 834 825 517 562 ...Usaremos, por simplicidad, variables binarias.
Datos$SEXFem=1*(Datos$SEX==2)
Datos$VITUSEYes=1*(Datos$VITUSE==1)
Datos$SEXFem=as.factor(Datos$SEXFem)
Datos$VITUSEYes=as.factor(Datos$VITUSEYes)
DatosRed=Datos[,c("BETAPLASMA", "AGE", "SEXFem", "VITUSEYes", "CALORIES", "FAT", "FIBER", "ALCOHOL", "CHOLESTEROL", "BETADIET", "VITUSE")]
summary(DatosRed)## BETAPLASMA AGE SEXFem VITUSEYes CALORIES FAT
## Min. : 0 Min. :19 0: 42 0:193 Min. : 445 Min. : 14
## 1st Qu.: 90 1st Qu.:39 1:273 1:122 1st Qu.:1338 1st Qu.: 54
## Median : 140 Median :48 Median :1667 Median : 73
## Mean : 190 Mean :50 Mean :1797 Mean : 77
## 3rd Qu.: 230 3rd Qu.:62 3rd Qu.:2100 3rd Qu.: 95
## Max. :1415 Max. :83 Max. :6662 Max. :236
## FIBER ALCOHOL CHOLESTEROL BETADIET VITUSE
## Min. : 3 Min. : 0 Min. : 38 Min. : 214 Min. :1.00
## 1st Qu.: 9 1st Qu.: 0 1st Qu.:155 1st Qu.:1116 1st Qu.:1.00
## Median :12 Median : 0 Median :206 Median :1802 Median :2.00
## Mean :13 Mean : 3 Mean :242 Mean :2186 Mean :1.97
## 3rd Qu.:16 3rd Qu.: 3 3rd Qu.:309 3rd Qu.:2836 3rd Qu.:3.00
## Max. :37 Max. :203 Max. :901 Max. :9642 Max. :3.00library(GGally)
ggpairs(DatosRed[,c(1,3,4)])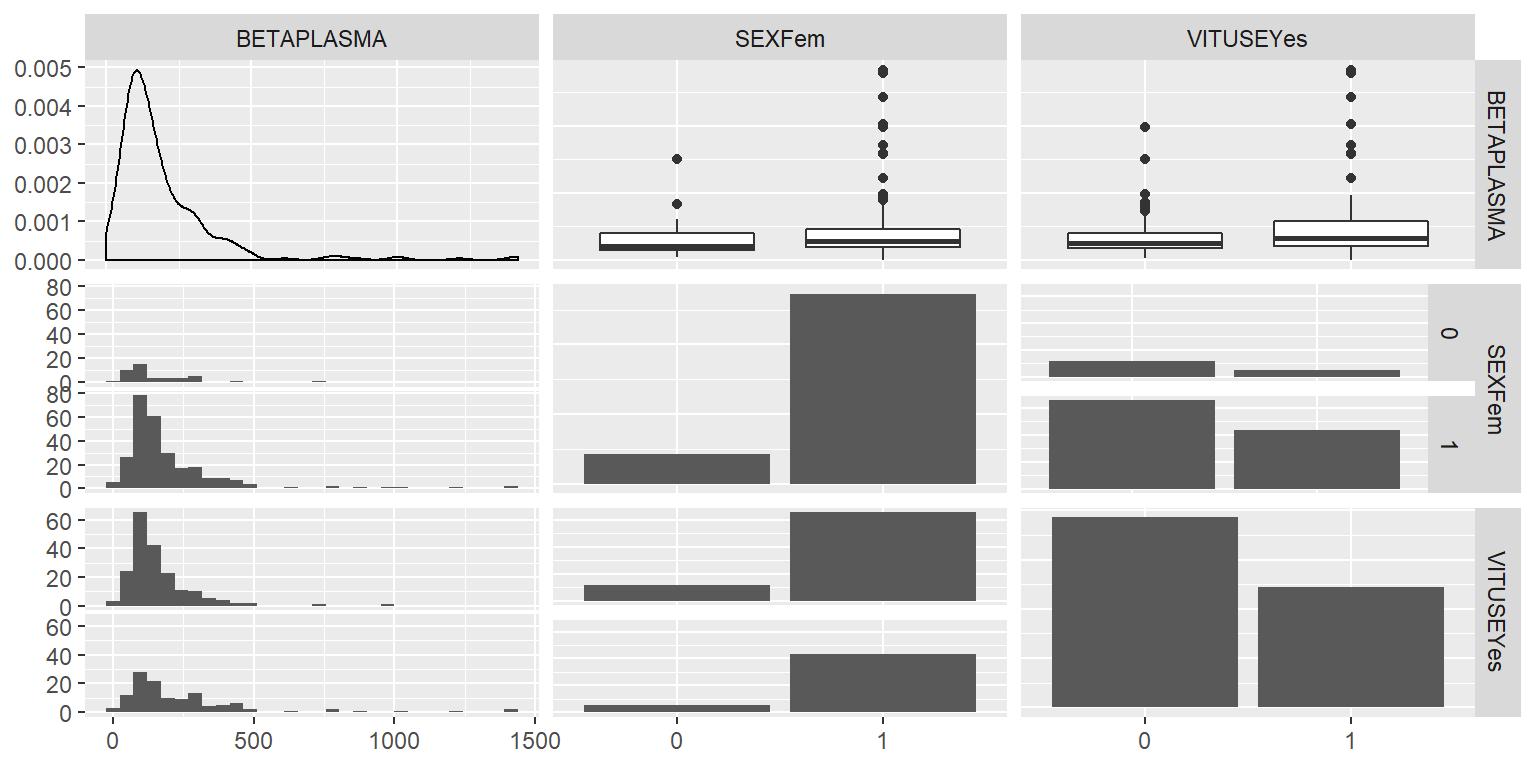
ggpairs(DatosRed[,c(1,2,5:10)])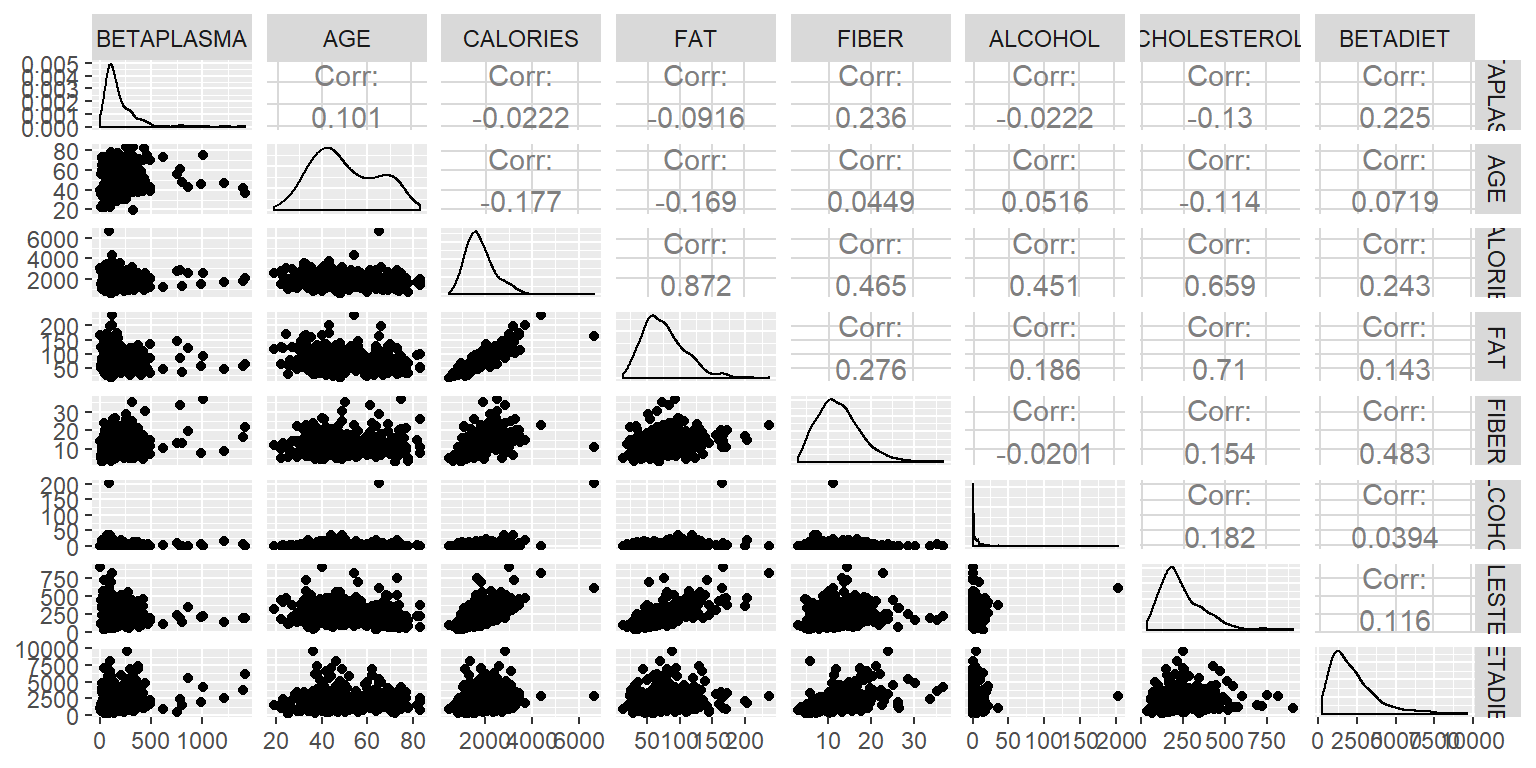
DatosRed[ DatosRed$ALCOHOL>100, ]DatosRed[ DatosRed$CALORIES>4000, ]DatosRed[ DatosRed$BETAPLASMA==0, ]DatosRed2=DatosRed[-c(62, 257), ]library(GGally)
ggpairs(DatosRed2[,c(1,3,4)])
ggpairs(DatosRed2[,c(1,2,5:10)])
DatosRed2$BETAPLASMAlog=log(DatosRed2$BETAPLASMA+10)
fit2=lm(BETAPLASMAlog~AGE+SEXFem+VITUSEYes+CALORIES+FAT+FIBER+ALCOHOL+CHOLESTEROL+BETADIET, data=DatosRed2)
summary(fit2)##
## Call:
## lm(formula = BETAPLASMAlog ~ AGE + SEXFem + VITUSEYes + CALORIES +
## FAT + FIBER + ALCOHOL + CHOLESTEROL + BETADIET, data = DatosRed2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.8298 -0.3546 -0.0217 0.3707 2.0218
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 4.27e+00 2.56e-01 16.69 <2e-16 ***
## AGE 5.05e-03 2.80e-03 1.81 0.0721 .
## SEXFem1 2.80e-01 1.20e-01 2.33 0.0206 *
## VITUSEYes1 2.24e-01 7.61e-02 2.94 0.0035 **
## CALORIES -1.69e-04 1.92e-04 -0.88 0.3799
## FAT 4.61e-04 3.03e-03 0.15 0.8792
## FIBER 3.49e-02 1.06e-02 3.30 0.0011 **
## ALCOHOL 8.05e-03 8.21e-03 0.98 0.3277
## CHOLESTEROL -4.18e-04 4.19e-04 -1.00 0.3193
## BETADIET 4.19e-05 2.83e-05 1.48 0.1386
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.64 on 303 degrees of freedom
## Multiple R-squared: 0.164, Adjusted R-squared: 0.14
## F-statistic: 6.62 on 9 and 303 DF, p-value: 1.21e-08drop1(fit2, test = "F")Ejemplo con una variable categórica con 3 niveles.
DatosRed2$VITUSE=as.factor(DatosRed2$VITUSE)
fit3=lm(BETAPLASMAlog~AGE+SEXFem+VITUSE+CALORIES+FAT+FIBER+ALCOHOL+CHOLESTEROL+BETADIET, data=DatosRed2)
summary(fit3)##
## Call:
## lm(formula = BETAPLASMAlog ~ AGE + SEXFem + VITUSE + CALORIES +
## FAT + FIBER + ALCOHOL + CHOLESTEROL + BETADIET, data = DatosRed2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.9640 -0.3569 -0.0446 0.3606 1.8759
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 4.53e+00 2.64e-01 17.15 < 2e-16 ***
## AGE 5.55e-03 2.77e-03 2.00 0.04648 *
## SEXFem1 2.37e-01 1.20e-01 1.98 0.04843 *
## VITUSE2 -7.68e-02 9.18e-02 -0.84 0.40324
## VITUSE3 -3.42e-01 8.63e-02 -3.96 9.3e-05 ***
## CALORIES -2.32e-04 1.91e-04 -1.21 0.22671
## FAT 1.47e-03 3.02e-03 0.49 0.62706
## FIBER 3.61e-02 1.04e-02 3.46 0.00062 ***
## ALCOHOL 9.85e-03 8.15e-03 1.21 0.22726
## CHOLESTEROL -4.40e-04 4.14e-04 -1.06 0.28903
## BETADIET 4.06e-05 2.79e-05 1.45 0.14761
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.63 on 302 degrees of freedom
## Multiple R-squared: 0.185, Adjusted R-squared: 0.158
## F-statistic: 6.88 on 10 and 302 DF, p-value: 1.09e-09drop1(fit3, test = "F")Para la variable VITUSE
library(multcomp)
K=matrix(c(0,0,0,1,0,0,0,0,0,0,0, 0,0,0,0,1,0,0,0,0,0,0), ncol=11, nrow=2, byrow=TRUE)
m=c(0, 0)
summary(glht(fit3, linfct=K, rhs=m),test=Ftest())##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## 1 == 0 -0.0768
## 2 == 0 -0.3419
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 8.33 2 302 3e-04Para categorías de la variable VITUSE
library(multcomp)
K=matrix(c(0,0,0,1,-1,0,0,0,0,0,0), ncol=11, nrow=1, byrow=TRUE)
m=c(0)
summary(glht(fit3, linfct=K, rhs=m),test=Ftest())##
## General Linear Hypotheses
##
## Linear Hypotheses:
## Estimate
## 1 == 0 0.265
##
## Global Test:
## F DF1 DF2 Pr(>F)
## 1 7.83 1 302 0.00547