Regresión lineal simple (4/4)
Gonzalo Pérez
5 de marzo de 2020
Análisis de residuales y verificación de supuestos del modelo.
En el modelo de regresión lineal simple se hacen principalmente los siguientes supuestos:
- Relación lineal entre la variable \(X\) y \(Y\).
- Errores independientes
- Errores con la misma varianza (homocedasticidad)
- Distribución normal de los errores
Otros problemas potenciales que se pueden encontrar son outliers (generalmente en la variable \(Y\)) y valores influyentes (en la variable \(X\)).
La mayoría de estos supuestos se realiza de forma gráfica y usando los residuales (o errores estimados) o adaptaciones de éstos.
Relación lineal entre la variable \(X\) y \(Y\).
Esta revisión se puede realizar en la regresión lineal simple directamente observando el scatterplot de los datos \(X\) y \(Y\).
library(ISLR)
Datos=read.csv("images/archivos/Advertising.csv", header=TRUE, sep="," )
require(ggplot2)
require(ggiraph)
require(ggiraphExtra)
ggPoints(aes(x=TV,y=sales),method="lm", data=Datos,interactive=TRUE)Sin embargo, es muy común usar también la gráfica de los residuales \(y_i-\hat{y}_i\) contra los valores ajustados \(\hat{y}_i\). El objeto obtenido al usar la función lm en R tiene preestablecidas varias gráficas que sirven para los análisis.
fit1=lm(sales~TV, data=Datos)
summary(fit1)##
## Call:
## lm(formula = sales ~ TV, data = Datos)
##
## Residuals:
## Min 1Q Median 3Q Max
## -8.386 -1.955 -0.191 2.067 7.212
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 7.03259 0.45784 15.4 <2e-16 ***
## TV 0.04754 0.00269 17.7 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.3 on 198 degrees of freedom
## Multiple R-squared: 0.612, Adjusted R-squared: 0.61
## F-statistic: 312 on 1 and 198 DF, p-value: <2e-16plot(fit1, 1)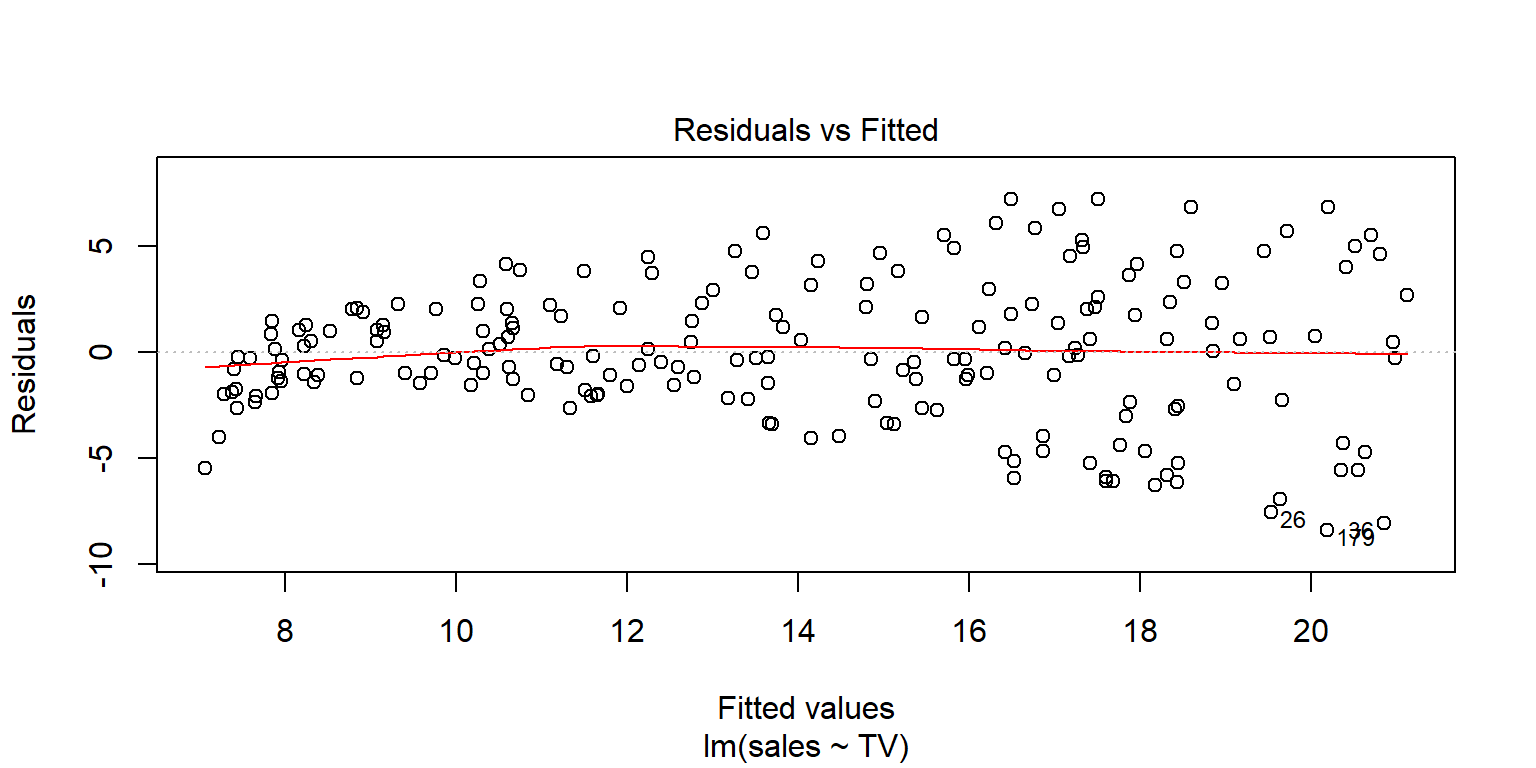
En este caso, lo ideal sería que los residuales no muestren ningún patrón con respecto a no seguir en promedio una línea horizontal con media cero. En el ejemplo, se puede observar que para los valores pequeños parece que hay una distorsión, aunque en general no es mucha, el patrón más marcado está sobre un crecimiento en la variabilidad de los residuales.
Opciones para arreglar la linealidad son considerar transformaciones a los datos, ya sea a la variable \(X\), a la variable \(Y\) o ambas.
Datos$Xprima=sqrt(Datos$TV)
fit2=lm(sales~Xprima, data=Datos)
summary(fit2)##
## Call:
## lm(formula = sales ~ Xprima, data = Datos)
##
## Residuals:
## Min 1Q Median 3Q Max
## -7.430 -1.953 -0.109 1.939 7.764
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 2.672 0.667 4.0 8.8e-05 ***
## Xprima 0.995 0.055 18.1 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.2 on 198 degrees of freedom
## Multiple R-squared: 0.623, Adjusted R-squared: 0.621
## F-statistic: 327 on 1 and 198 DF, p-value: <2e-16plot(fit2, 1)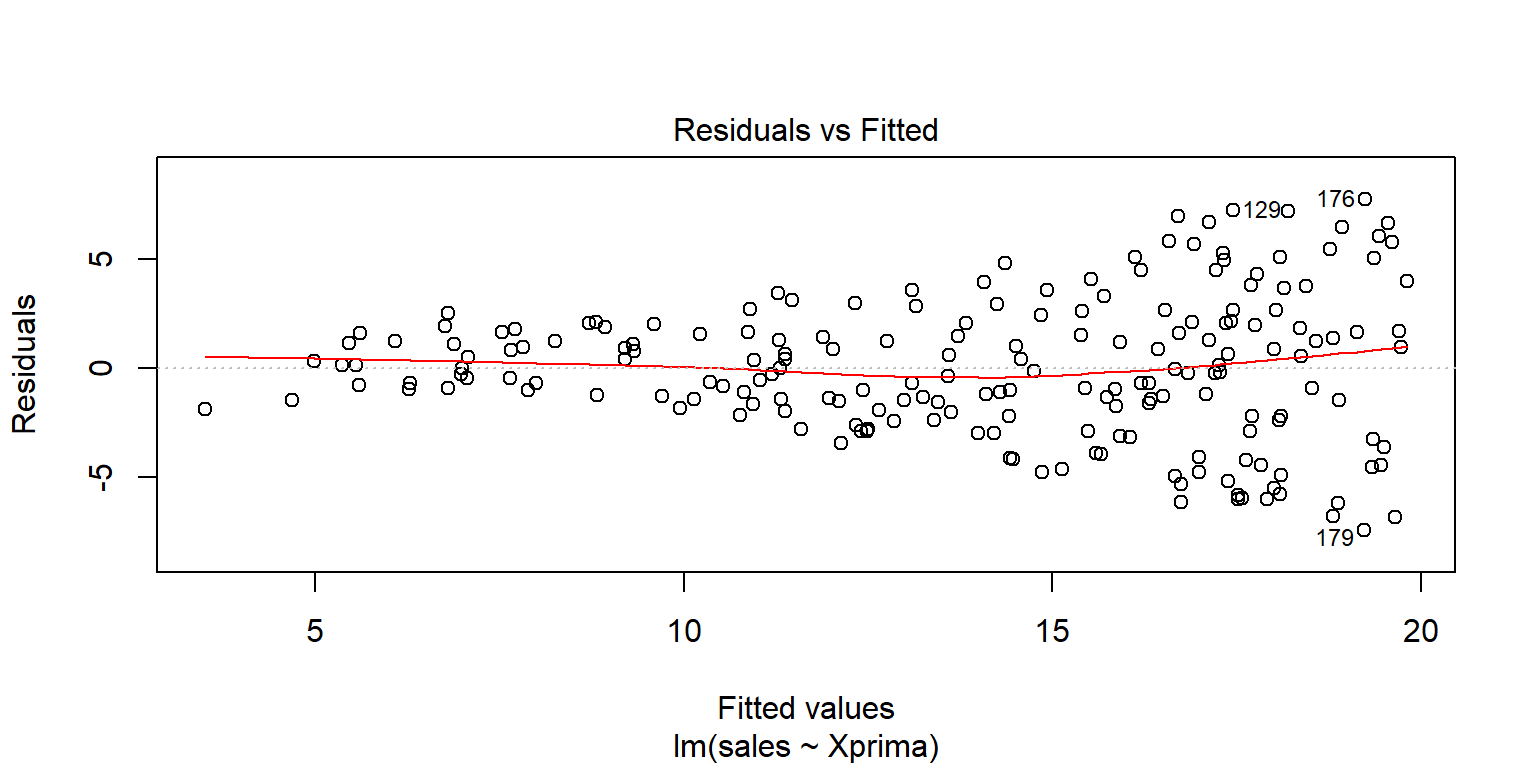
ggPoints(aes(x=Xprima,y=sales),method="lm", data=Datos,interactive=TRUE)Lo anterior parece arreglar la linealidad, pero no la variabilidad no constante en \(Y\).
Una tranformación polinomial también podría ser una opción, aunque eso ya es regresión lineal múltiple
Datos$Xprima2=Datos$TV^2
Datos$Xprima3=Datos$TV^3
fit3=lm(sales~TV+Xprima2+Xprima3, data=Datos)
summary(fit3)##
## Call:
## lm(formula = sales ~ TV + Xprima2 + Xprima3, data = Datos)
##
## Residuals:
## Min 1Q Median 3Q Max
## -7.97 -1.89 -0.09 2.02 7.38
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 5.42e+00 8.64e-01 6.27 2.2e-09 ***
## TV 9.64e-02 2.58e-02 3.74 0.00024 ***
## Xprima2 -3.15e-04 2.02e-04 -1.56 0.12056
## Xprima3 5.57e-07 4.49e-07 1.24 0.21652
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.2 on 196 degrees of freedom
## Multiple R-squared: 0.622, Adjusted R-squared: 0.616
## F-statistic: 108 on 3 and 196 DF, p-value: <2e-16plot(fit3, 1)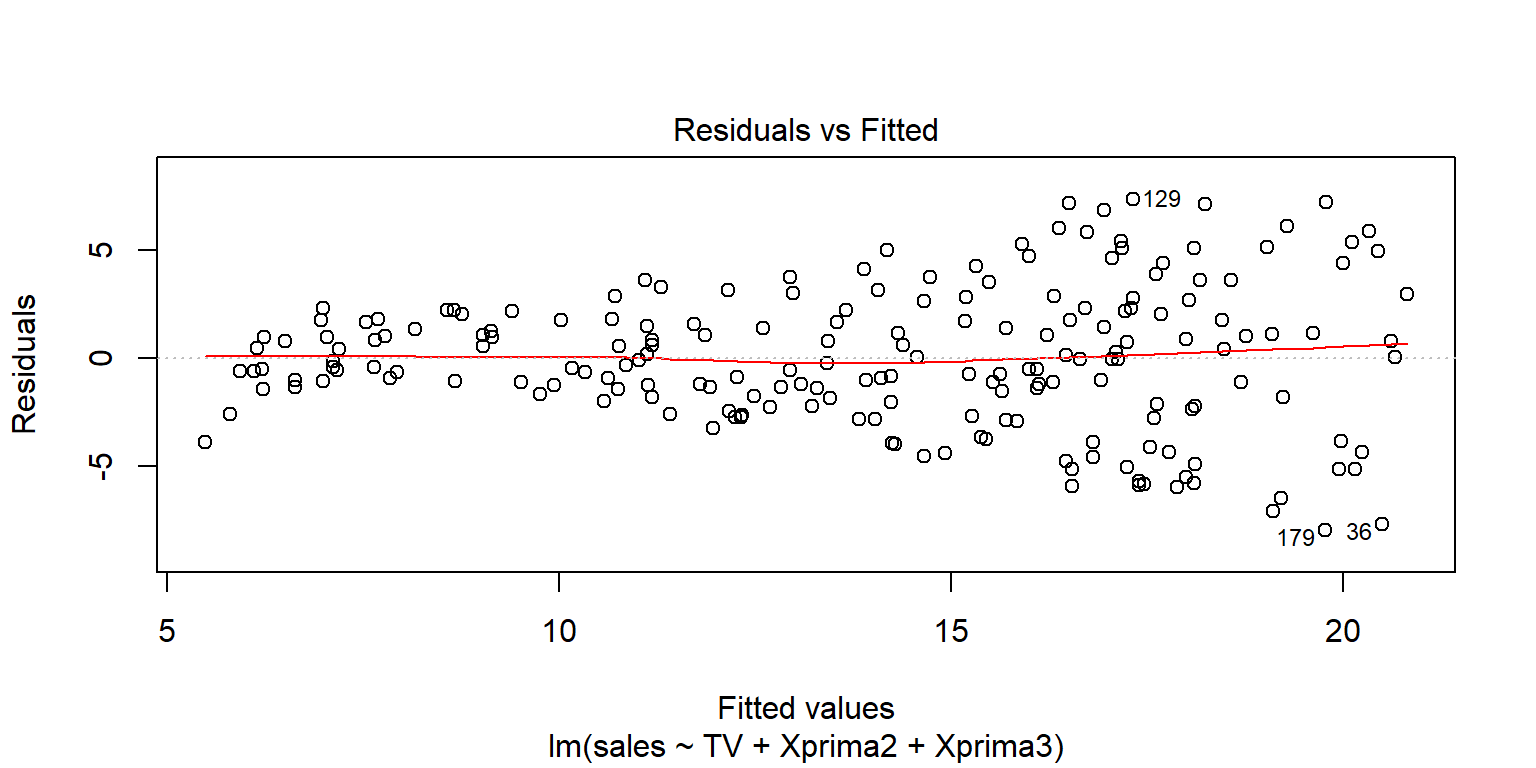
ggplot(data=Datos, aes(x=TV,y=sales))+
geom_smooth(method="lm", se=FALSE, colour="red", formula=y~x)+
geom_smooth(method="lm", se=FALSE, colour="blue", formula=y~x+I(x^2)+I(x^3))+
geom_point(colour="black")
A veces es conveniente transformar la variable \(Y\), con suerte eso eliminaría la varianza no constante.
library(car)
fit1Ymod=boxCox(fit1, plotit=TRUE)
Datos$Yprima=( (Datos$sales)^(.5)-1 )/.5
Datos$Yprima2=bcPower(Datos$sales, .5)
fit4=lm(Yprima~TV, data=Datos)
summary(fit4)##
## Call:
## lm(formula = Yprima ~ TV, data = Datos)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.8991 -0.5049 0.0234 0.6365 1.7001
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.419720 0.121136 28.2 <2e-16 ***
## TV 0.013156 0.000712 18.5 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.86 on 198 degrees of freedom
## Multiple R-squared: 0.633, Adjusted R-squared: 0.631
## F-statistic: 342 on 1 and 198 DF, p-value: <2e-16plot(fit4, 1)
datonuevos= data.frame(TV=seq(0, 300, by=.2), Yprima=predict(fit4, newdata=data.frame(TV=seq(0, 300, by=.2) ) ))
datonuevos$sales=(.5*(datonuevos$Yprima)+1)^2
ggPoints(aes(x=TV,y=Yprima),method="lm", data=Datos,interactive=TRUE)ggplot(data=Datos, aes(x=TV,y=sales))+
geom_smooth(method="lm", se=FALSE, colour="red", formula=y~x)+
geom_smooth(method="lm", se=FALSE, colour="blue", formula=y~I(x^(.5)))+
geom_point(colour="black")+
geom_line(data= datonuevos, colour="green" )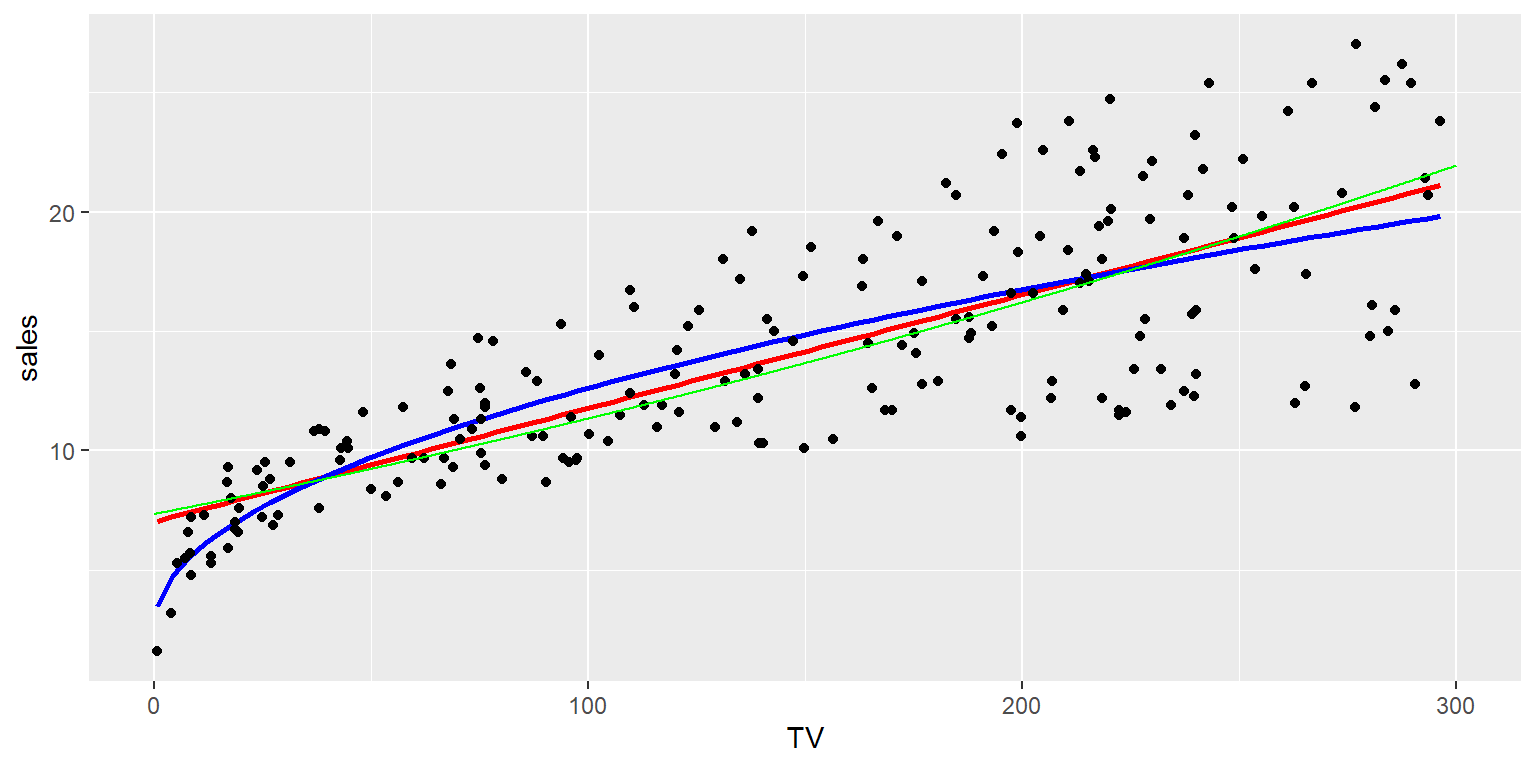
boxTidwell(Yprima~TV, data=Datos)## MLE of lambda Score Statistic (z) Pr(>|z|)
## 0.3 -4 6e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## iterations = 4Datos$Xprimab=Datos$TV^(.3)
fit5=lm(Yprima~Xprimab, data=Datos)
summary(fit5)##
## Call:
## lm(formula = Yprima ~ Xprimab, data = Datos)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.8267 -0.5371 0.0111 0.5710 1.6940
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 0.5526 0.2416 2.29 0.023 *
## Xprimab 1.1373 0.0556 20.46 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.81 on 198 degrees of freedom
## Multiple R-squared: 0.679, Adjusted R-squared: 0.677
## F-statistic: 418 on 1 and 198 DF, p-value: <2e-16plot(fit5, 1)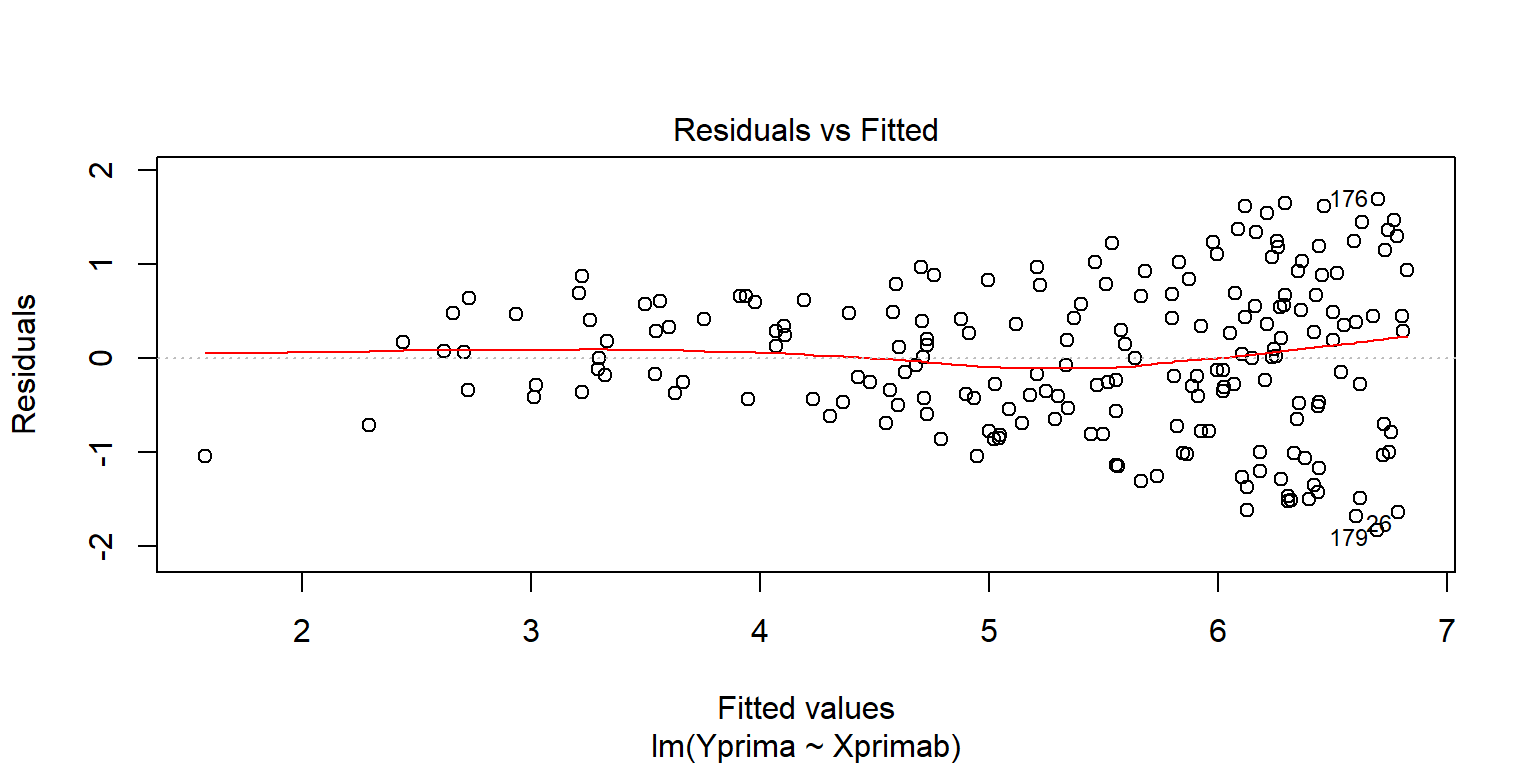
datonuevos= data.frame(Xprimab=seq(0, 5.5, by=.2), Yprima=predict(fit5, newdata=data.frame(Xprimab=seq(0, 5.5, by=.2) ) ))
datonuevos$TV=(datonuevos$Xprimab)^(1/.3)
datonuevos$sales=(.5*(datonuevos$Yprima)+1)^2
ggPoints(aes(x=Xprimab,y=Yprima),method="lm", data=Datos,interactive=TRUE)ggplot(data=Datos, aes(x=TV,y=sales))+
geom_smooth(method="lm", se=FALSE, colour="red", formula=y~x)+
geom_smooth(method="lm", se=FALSE, colour="blue", formula=y~I(x^(.5)))+
geom_point(colour="black")+
geom_line(data= datonuevos, colour="green" )
Homocedasticidad
Se usa la gráfica de los residuales estandarizados contra los valores ajustados \(\hat{y}_i\). Se busca que los errores se distribuyan sin ningún patrón
plot(fit1, 3)
plot(fit2, 3)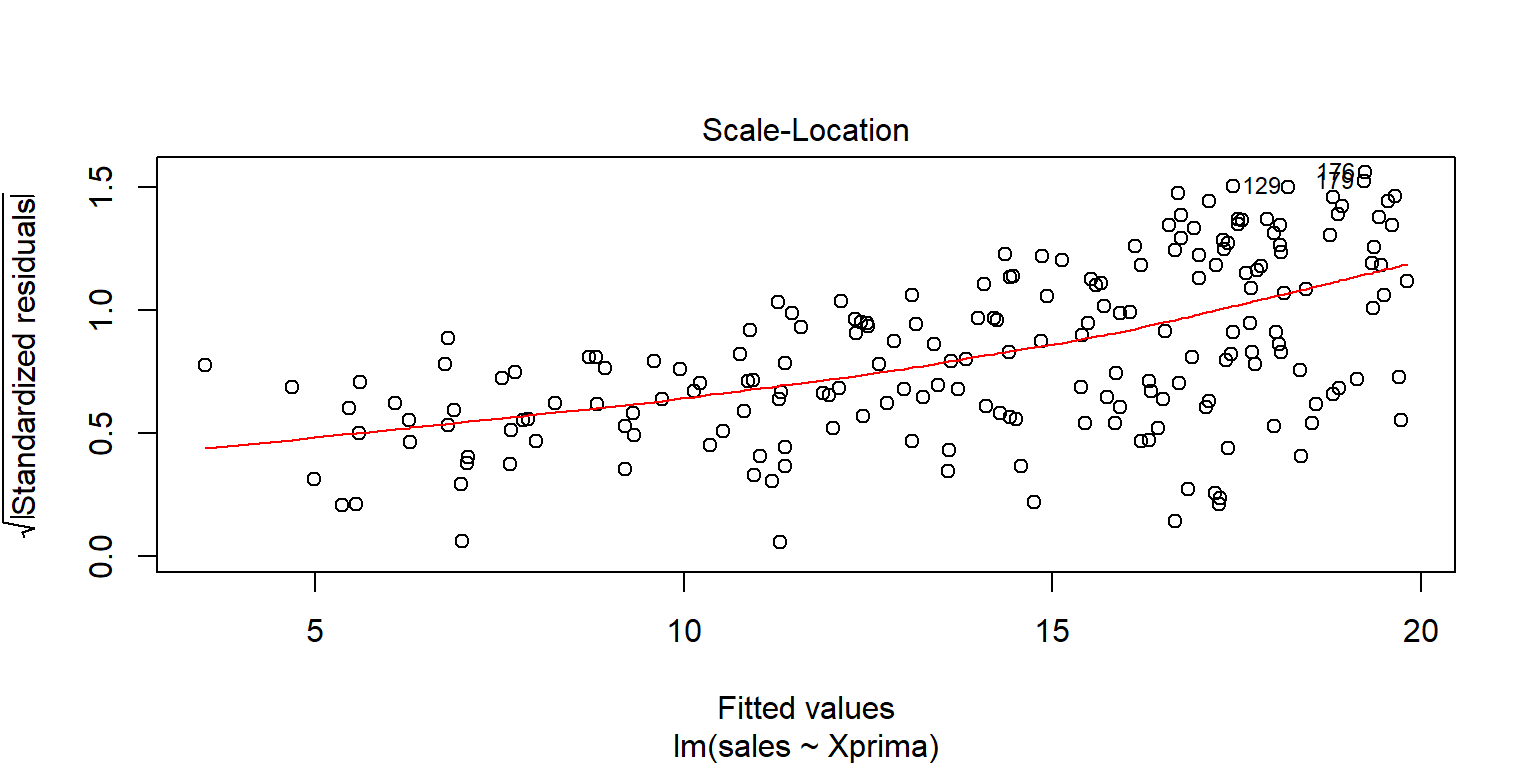
Existen algunas pruebas que pueden ayudar.
- Breush Pagan Test. \(H_0\): La varianza de los residuales no depende de las variables en el modelo, es decir, es constante.
library(lmtest)
lmtest::bptest(fit1)##
## studentized Breusch-Pagan test
##
## data: fit1
## BP = 48, df = 1, p-value = 4e-12lmtest::bptest(fit2)##
## studentized Breusch-Pagan test
##
## data: fit2
## BP = 53, df = 1, p-value = 3e-13- Score Test
library(car)
car::ncvTest(fit1)## Non-constant Variance Score Test
## Variance formula: ~ fitted.values
## Chisquare = 43, Df = 1, p = 6e-11car::ncvTest(fit2)## Non-constant Variance Score Test
## Variance formula: ~ fitted.values
## Chisquare = 46, Df = 1, p = 1e-11Errores independientes
library(broom)
Datosfit1=augment(fit1)
head(Datosfit1)## # A tibble: 6 x 9
## sales TV .fitted .se.fit .resid .hat .sigma .cooksd .std.resid
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 22.1 230. 18.0 0.321 4.13 0.00970 3.25 0.00794 1.27
## 2 10.4 44.5 9.15 0.359 1.25 0.0122 3.27 0.000920 0.387
## 3 9.3 17.2 7.85 0.419 1.45 0.0165 3.27 0.00169 0.449
## 4 18.5 152. 14.2 0.231 4.27 0.00501 3.25 0.00434 1.31
## 5 12.9 181. 15.6 0.248 -2.73 0.00578 3.26 0.00205 -0.839
## 6 7.2 8.7 7.45 0.438 -0.246 0.0180 3.27 0.0000534 -0.0762acf(Datosfit1$.std.resid)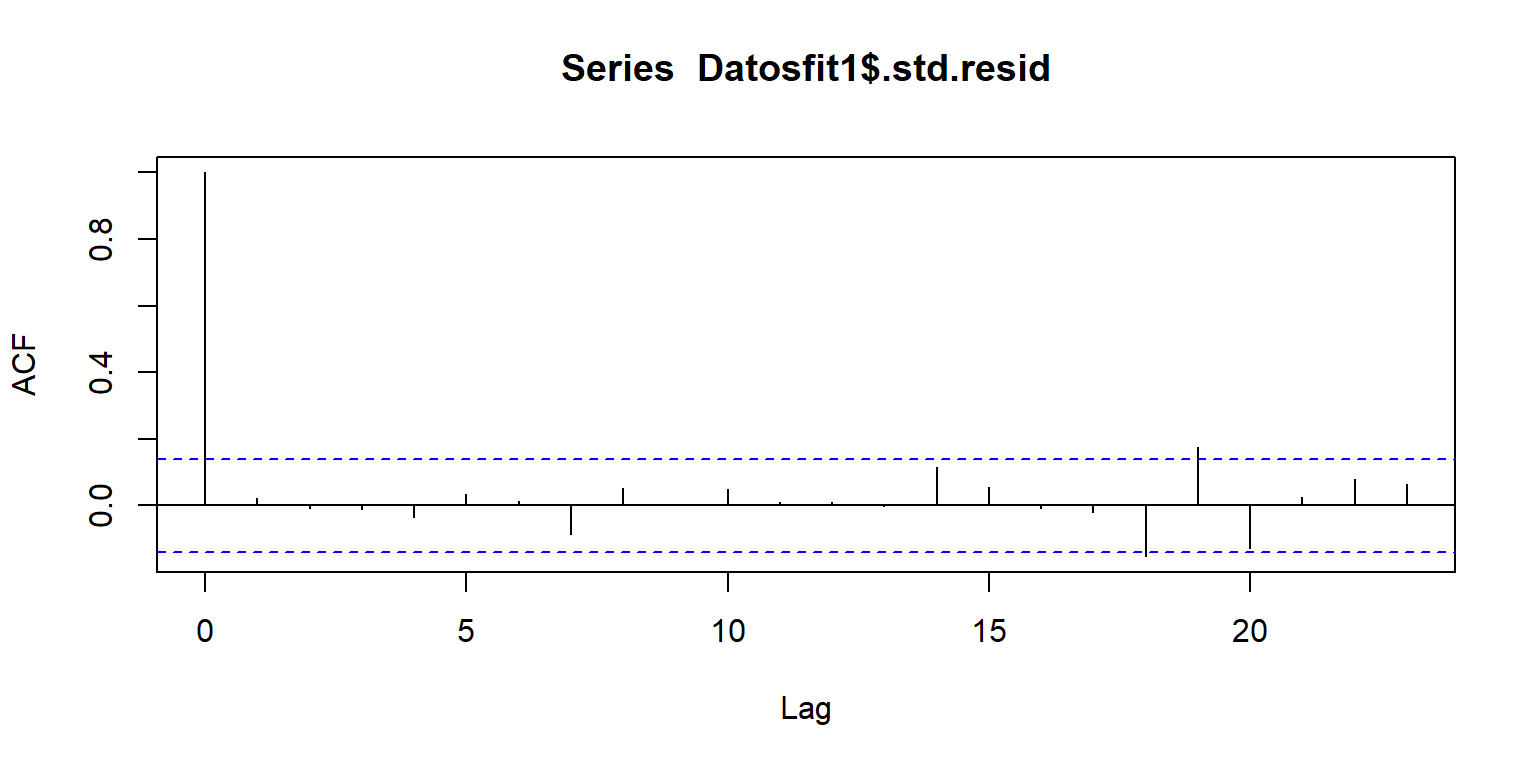
Algunas pruebas
Prueba de rachas. \(H_0\) los datos son aleatorios
library(lawstat)
lawstat::runs.test(Datosfit1$.std.resid)##
## Runs Test - Two sided
##
## data: Datosfit1$.std.resid
## Standardized Runs Statistic = -1, p-value = 0.3library(randtests)
randtests::runs.test(Datosfit1$.std.resid)##
## Runs Test
##
## data: Datosfit1$.std.resid
## statistic = -1, runs = 94, n1 = 100, n2 = 100, n = 200, p-value = 0.3
## alternative hypothesis: nonrandomnessPrueba Durbin-Watson. \(H_0\) los errores no están autocorrelacionados.
lmtest::dwtest(fit1)##
## Durbin-Watson test
##
## data: fit1
## DW = 2, p-value = 0.3
## alternative hypothesis: true autocorrelation is greater than 0Normalidad de los residuales
Por lo general se revisa con una qq-plot de los errores estandarizados. lm tiene una gráfica directa. Se desea que los puntos estén sobre la línea recta dibujada.
plot(fit1, 2)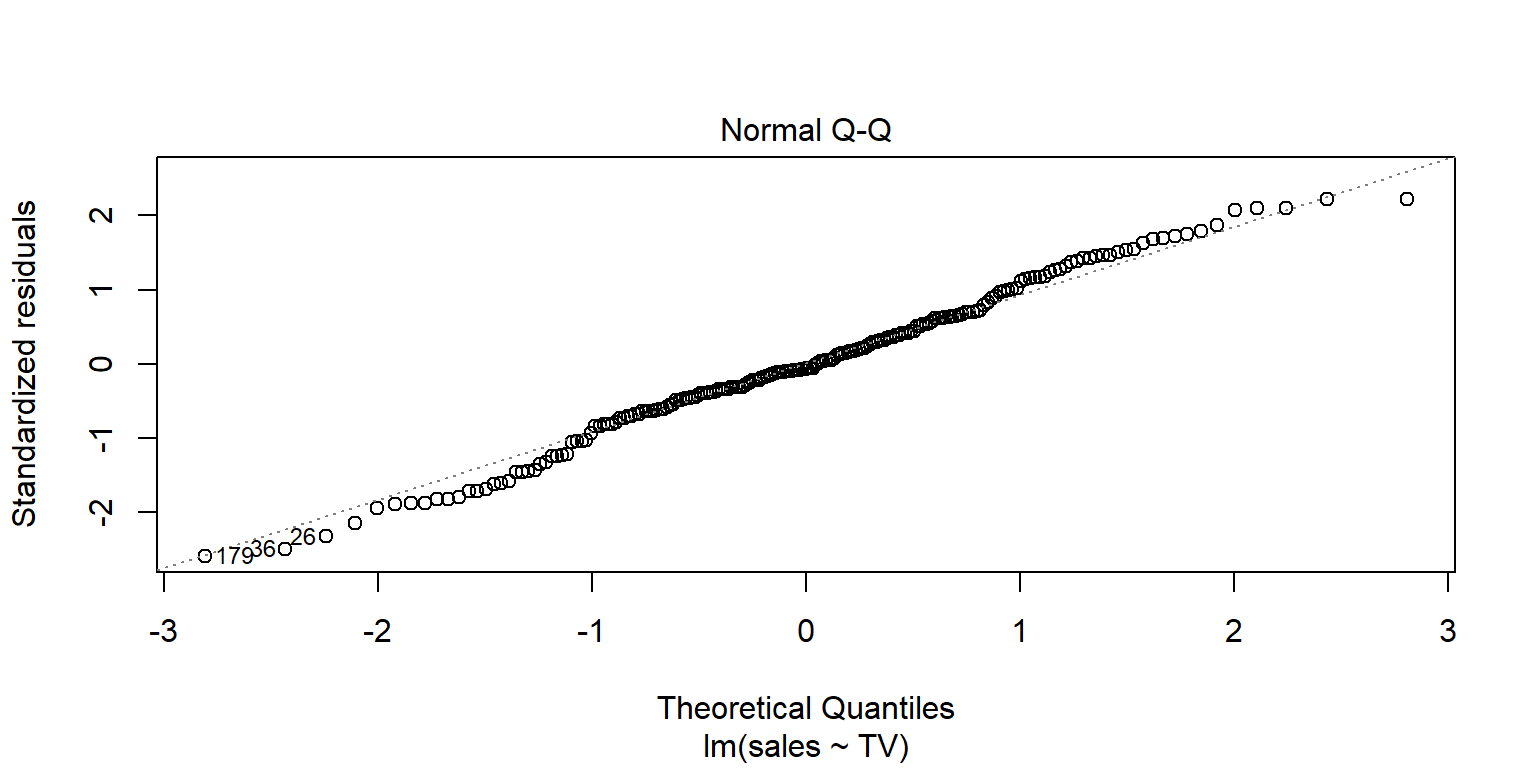
Algunos test. \(H_0\): los valores son normales.
shapiro.test(Datosfit1$.std.resid)##
## Shapiro-Wilk normality test
##
## data: Datosfit1$.std.resid
## W = 1, p-value = 0.2library(nortest)
nortest::lillie.test(Datosfit1$.std.resid)##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: Datosfit1$.std.resid
## D = 0.04, p-value = 0.5library(normtest)
normtest::jb.norm.test(Datosfit1$.std.resid)##
## Jarque-Bera test for normality
##
## data: Datosfit1$.std.resid
## JB = 0.7, p-value = 0.7Outliers y valores influyentes
Outliers se pueden observar con la gráfica de residuales estandarizados. En general, se consideran que los residuales más grandes de 2 (3) podrían ser outliers.
Existen algunas observaciones cuyo valor es extremo en la variable predictora. Se recomienda revisar la estadística leverage. En general, si esta estadística es superior a \(4/n\) se dice que tiene un valor muy grande en \(X\).
Las observaciones influyentes son aquellas que al eliminarlas pueden alterar los resultados de la regresión. Una metrica para analizar estos valores se conoce como Cook’s distance. Una regla empírica es considerar como observación influyente si esta distancia excede \(4/(n-2)\).
En R, existe una gráfica que resume lo anterior
plot(fit1, 5)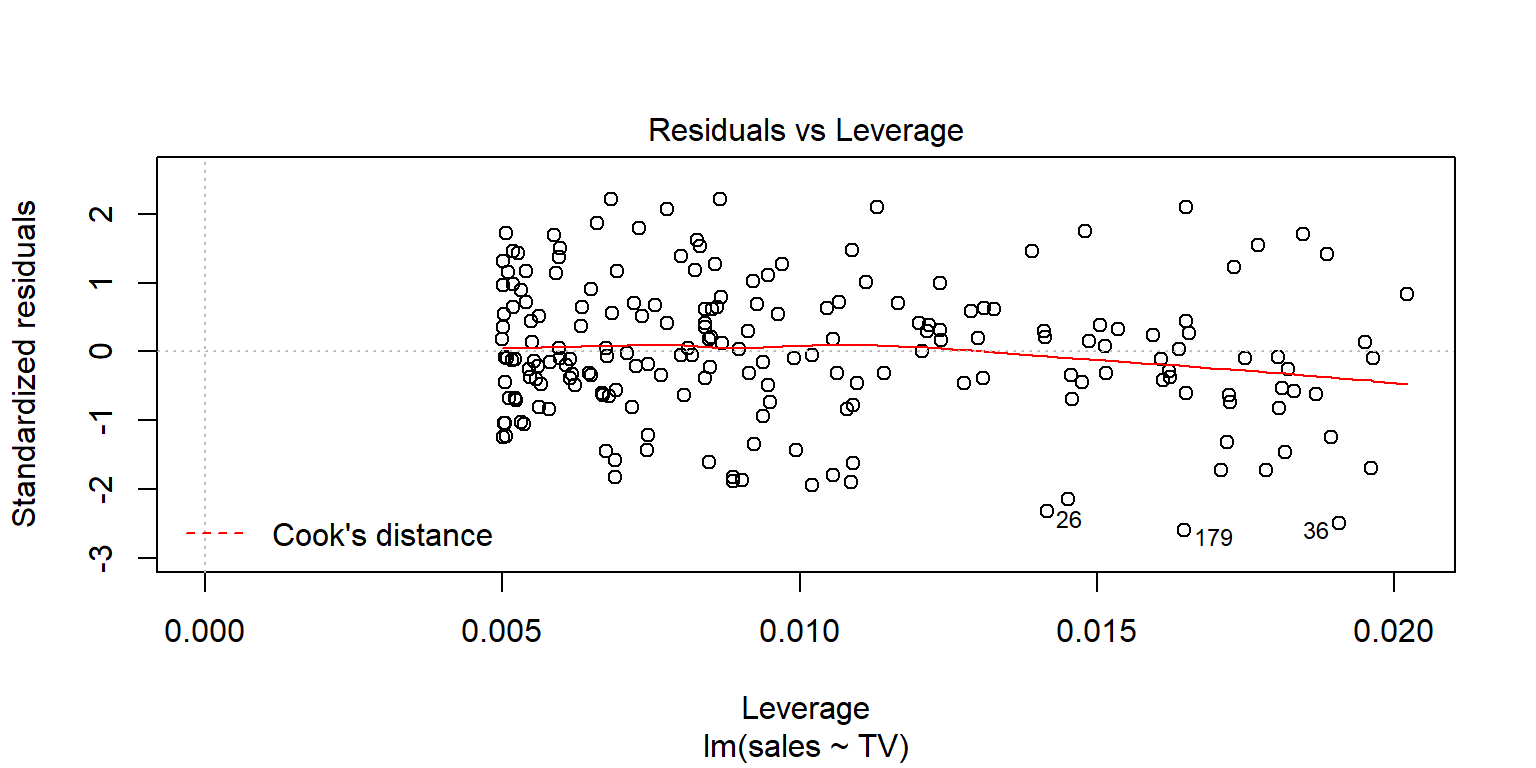
plot(fit1, 4)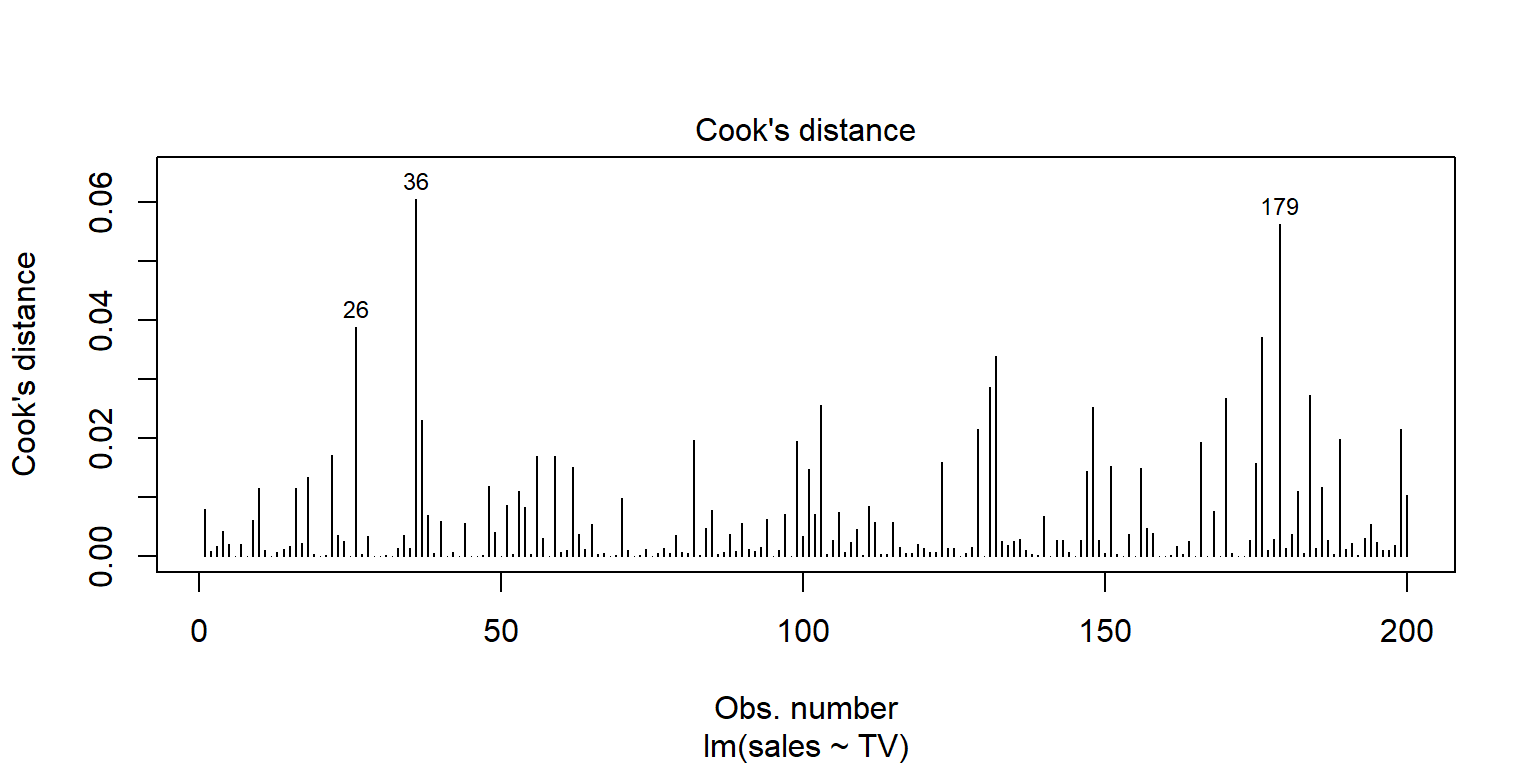
Otro paquete
library(olsrr)
ols_plot_diagnostics(fit1)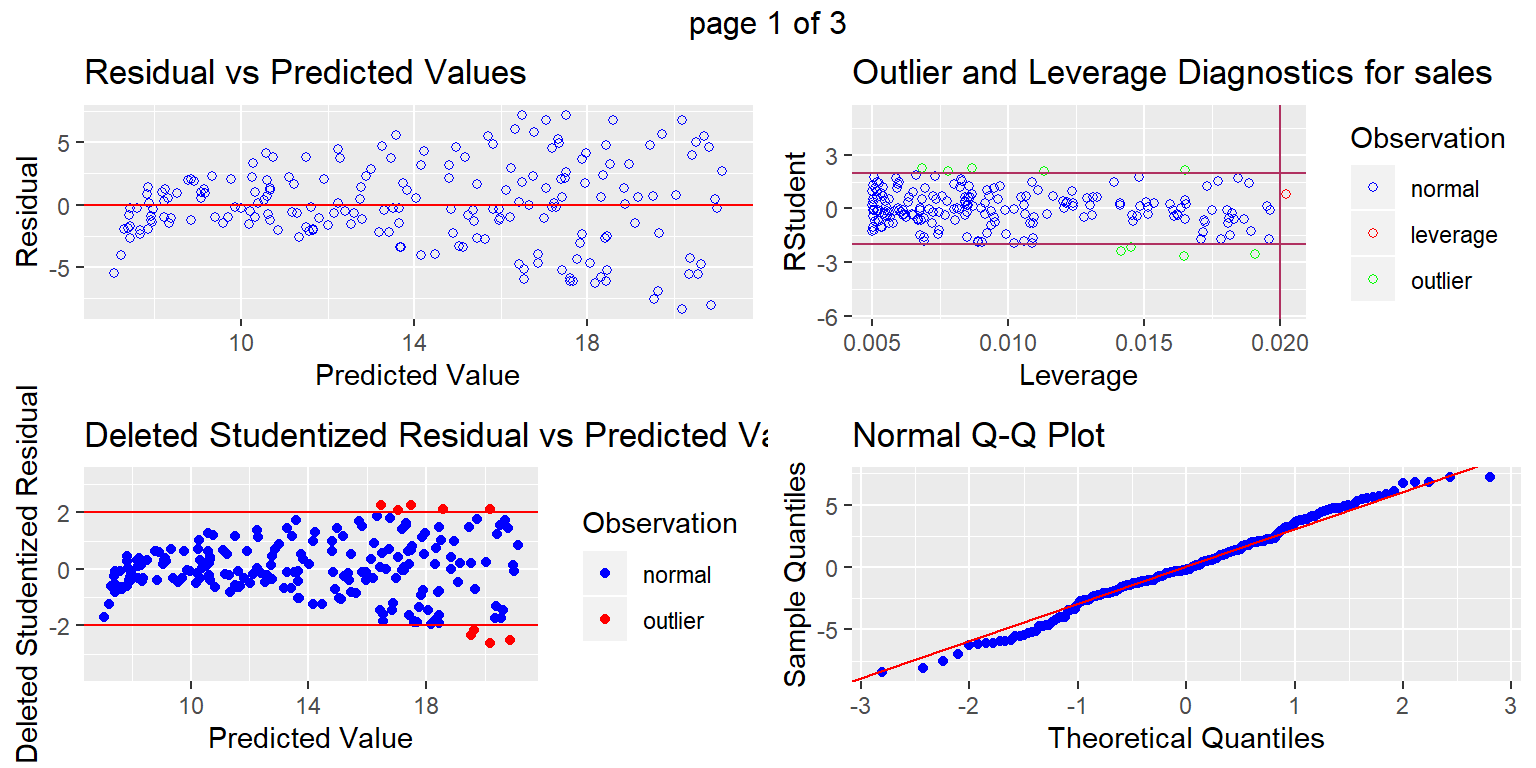
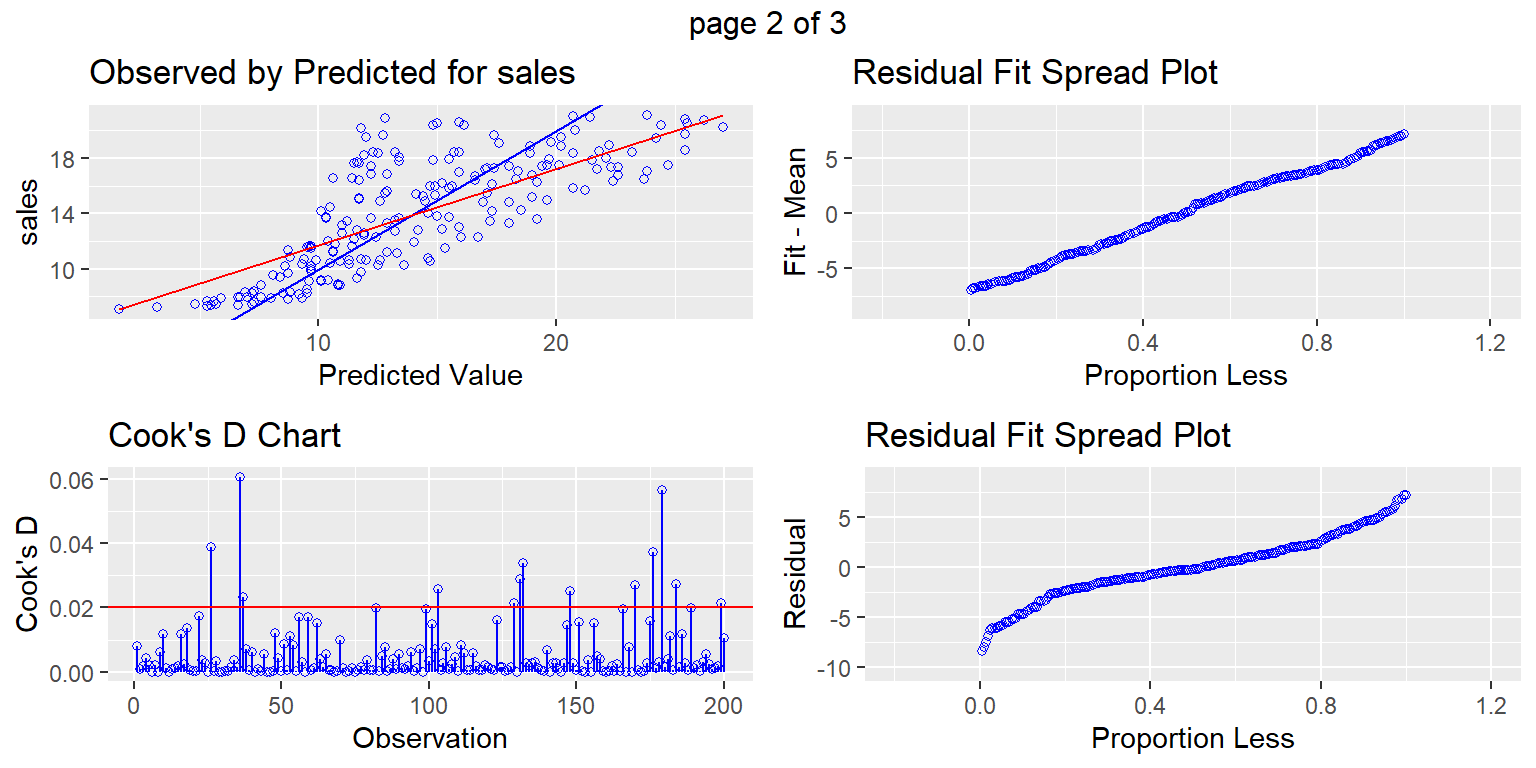

Regresión ponderada
Datos$Xprima=sqrt(Datos$TV)
fit2=lm(sales~Xprima, data=Datos)
summary(fit2)##
## Call:
## lm(formula = sales ~ Xprima, data = Datos)
##
## Residuals:
## Min 1Q Median 3Q Max
## -7.430 -1.953 -0.109 1.939 7.764
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 2.672 0.667 4.0 8.8e-05 ***
## Xprima 0.995 0.055 18.1 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.2 on 198 degrees of freedom
## Multiple R-squared: 0.623, Adjusted R-squared: 0.621
## F-statistic: 327 on 1 and 198 DF, p-value: <2e-16plot(fit2, 1)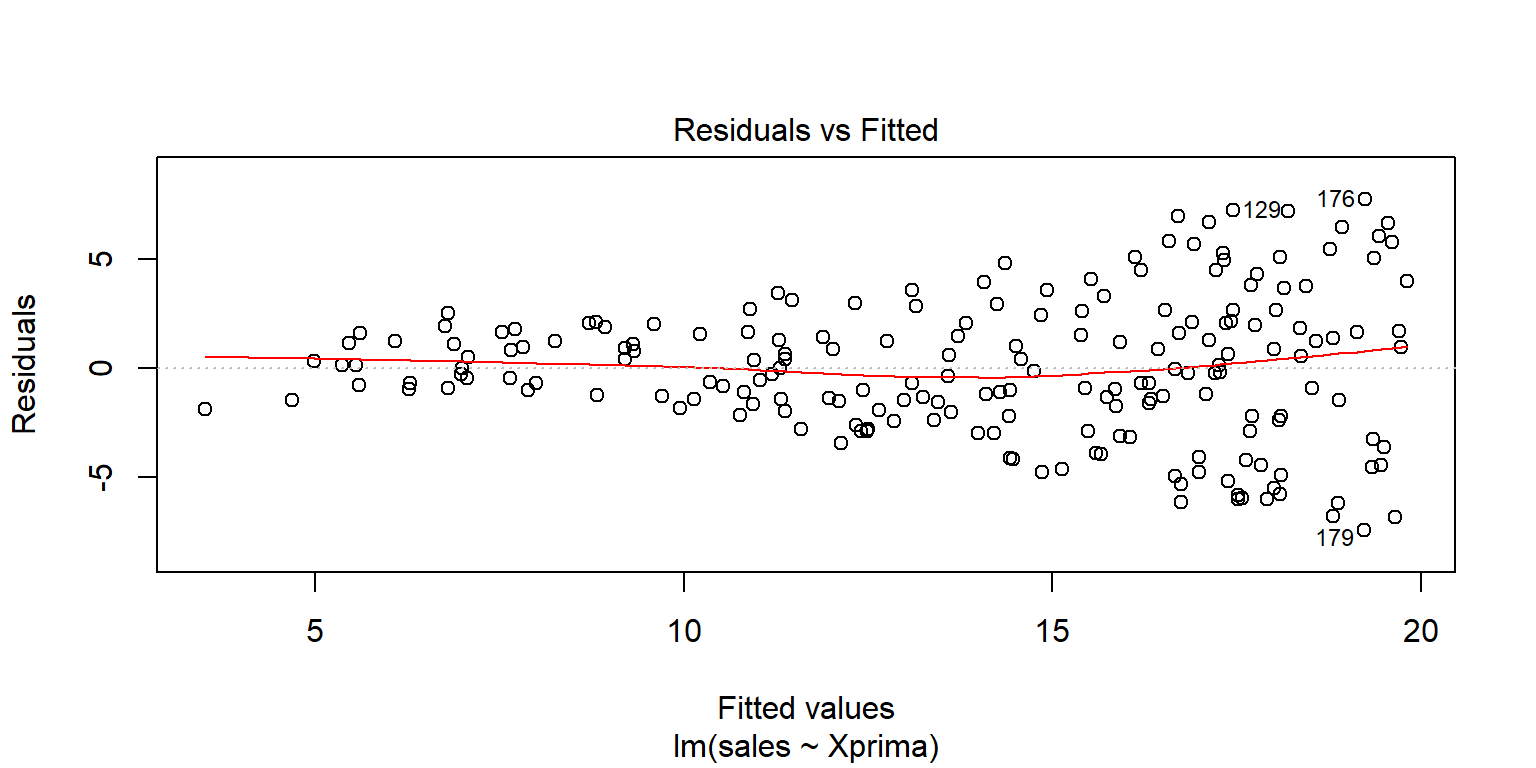
ggPoints(aes(x=Xprima,y=sales),method="lm", data=Datos,interactive=TRUE)Datosfit2=augment(fit2)
head(Datosfit2)## # A tibble: 6 x 9
## sales Xprima .fitted .se.fit .resid .hat .sigma .cooksd .std.resid
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 22.1 15.2 17.8 0.308 4.33 0.00917 3.21 0.00848 1.35
## 2 10.4 6.67 9.31 0.346 1.09 0.0116 3.22 0.000679 0.341
## 3 9.3 4.15 6.80 0.459 2.50 0.0205 3.22 0.00645 0.786
## 4 18.5 12.3 14.9 0.233 3.58 0.00524 3.21 0.00328 1.12
## 5 12.9 13.4 16.1 0.253 -3.16 0.00623 3.21 0.00305 -0.986
## 6 7.2 2.95 5.61 0.518 1.59 0.0260 3.22 0.00336 0.502ggPoints(aes(x=Xprima,y=.resid),smooth=FALSE, data=Datosfit2,interactive=TRUE)Datosfit2$res2=Datosfit2$.resid^2
ggPoints(aes(x=Xprima,y=res2),smooth=FALSE, data=Datosfit2,interactive=TRUE)fit2res=lm(res2~Xprima, data=Datosfit2)
summary(fit2res)##
## Call:
## lm(formula = res2 ~ Xprima, data = Datosfit2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -18.87 -8.11 -1.28 4.61 41.28
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -8.888 2.393 -3.71 0.00026 ***
## Xprima 1.675 0.197 8.49 4.9e-15 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 12 on 198 degrees of freedom
## Multiple R-squared: 0.267, Adjusted R-squared: 0.263
## F-statistic: 72.1 on 1 and 198 DF, p-value: 4.87e-15fit2res2=lm(res2~Xprima+I(Xprima^2), data=Datosfit2)
summary(fit2res2)##
## Call:
## lm(formula = res2 ~ Xprima + I(Xprima^2), data = Datosfit2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -24.21 -6.21 -0.84 4.27 37.14
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 6.3385 4.6805 1.35 0.17721
## Xprima -1.9660 0.9910 -1.98 0.04866 *
## I(Xprima^2) 0.1788 0.0478 3.74 0.00024 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 11 on 197 degrees of freedom
## Multiple R-squared: 0.316, Adjusted R-squared: 0.309
## F-statistic: 45.4 on 2 and 197 DF, p-value: <2e-16fit2res3=lm(res2~-1+Xprima+I(Xprima^2), data=Datosfit2)
summary(fit2res3)##
## Call:
## lm(formula = res2 ~ -1 + Xprima + I(Xprima^2), data = Datosfit2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -23.04 -6.86 0.49 4.14 37.99
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## Xprima -0.7014 0.3325 -2.11 0.036 *
## I(Xprima^2) 0.1226 0.0237 5.17 5.6e-07 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 11 on 198 degrees of freedom
## Multiple R-squared: 0.564, Adjusted R-squared: 0.559
## F-statistic: 128 on 2 and 198 DF, p-value: <2e-16Una opción es tomar como pesos \(w=1/(x)\)
fit2pond <- lm(sales~Xprima,weights = 1/( Xprima ), data=Datos)
summary(fit2pond)##
## Call:
## lm(formula = sales ~ Xprima, data = Datos, weights = 1/(Xprima))
##
## Weighted Residuals:
## Min 1Q Median 3Q Max
## -1.9096 -0.6108 -0.0376 0.6791 1.8839
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 2.5013 0.3831 6.53 5.4e-10 ***
## Xprima 1.0104 0.0384 26.29 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.89 on 198 degrees of freedom
## Multiple R-squared: 0.777, Adjusted R-squared: 0.776
## F-statistic: 691 on 1 and 198 DF, p-value: <2e-16datonuevos= data.frame(Xprima=seq(0, 17, by=.2), sales=predict(fit2pond, newdata=data.frame(Xprima=seq(0, 17, by=.2) ) ))
datonuevos$TV=(datonuevos$Xprima)^(2)
ggplot(data=Datos, aes(x=TV,y=sales))+
geom_point(colour="black")+
geom_line(data= datonuevos, colour="green" )
plot(fit2pond)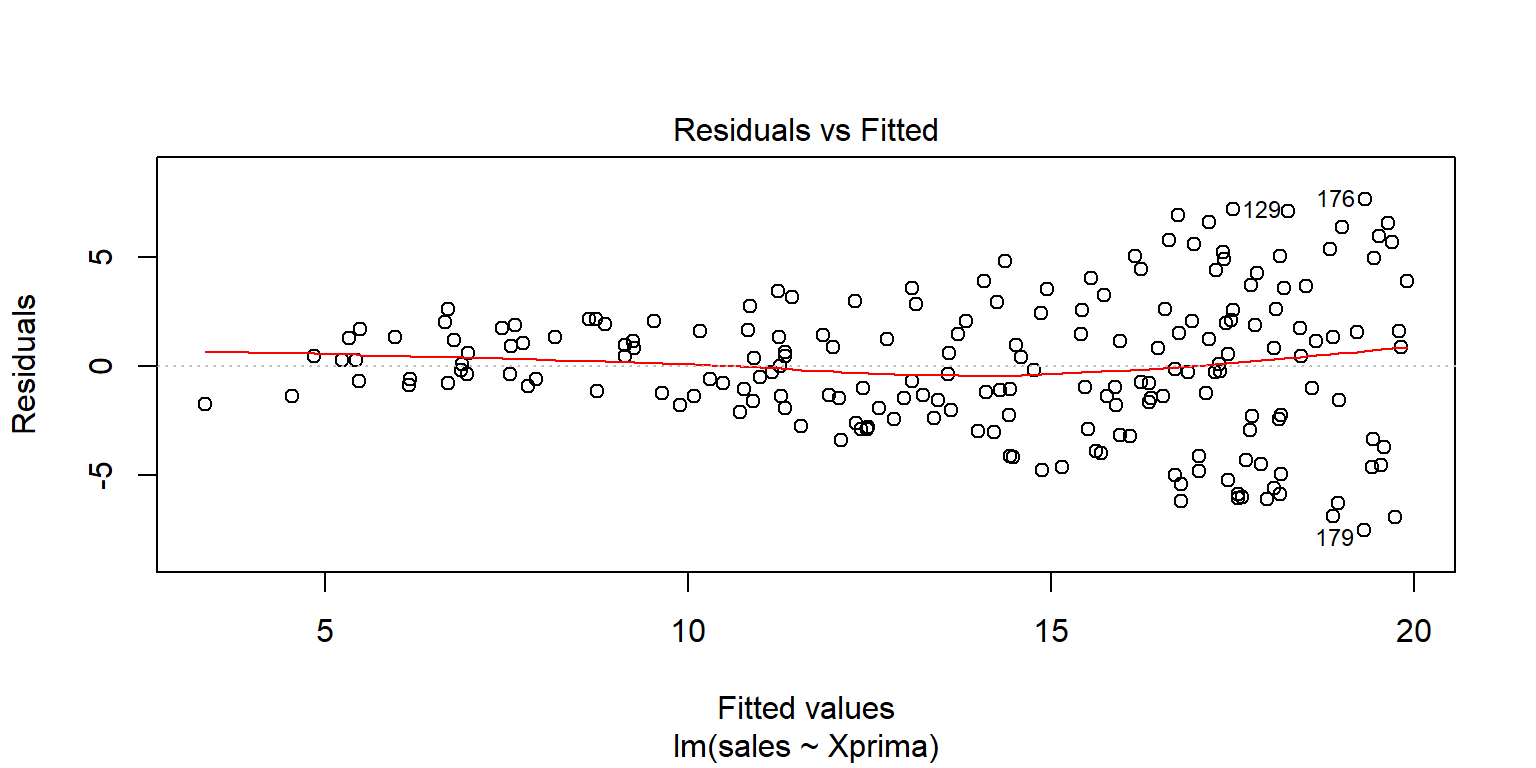

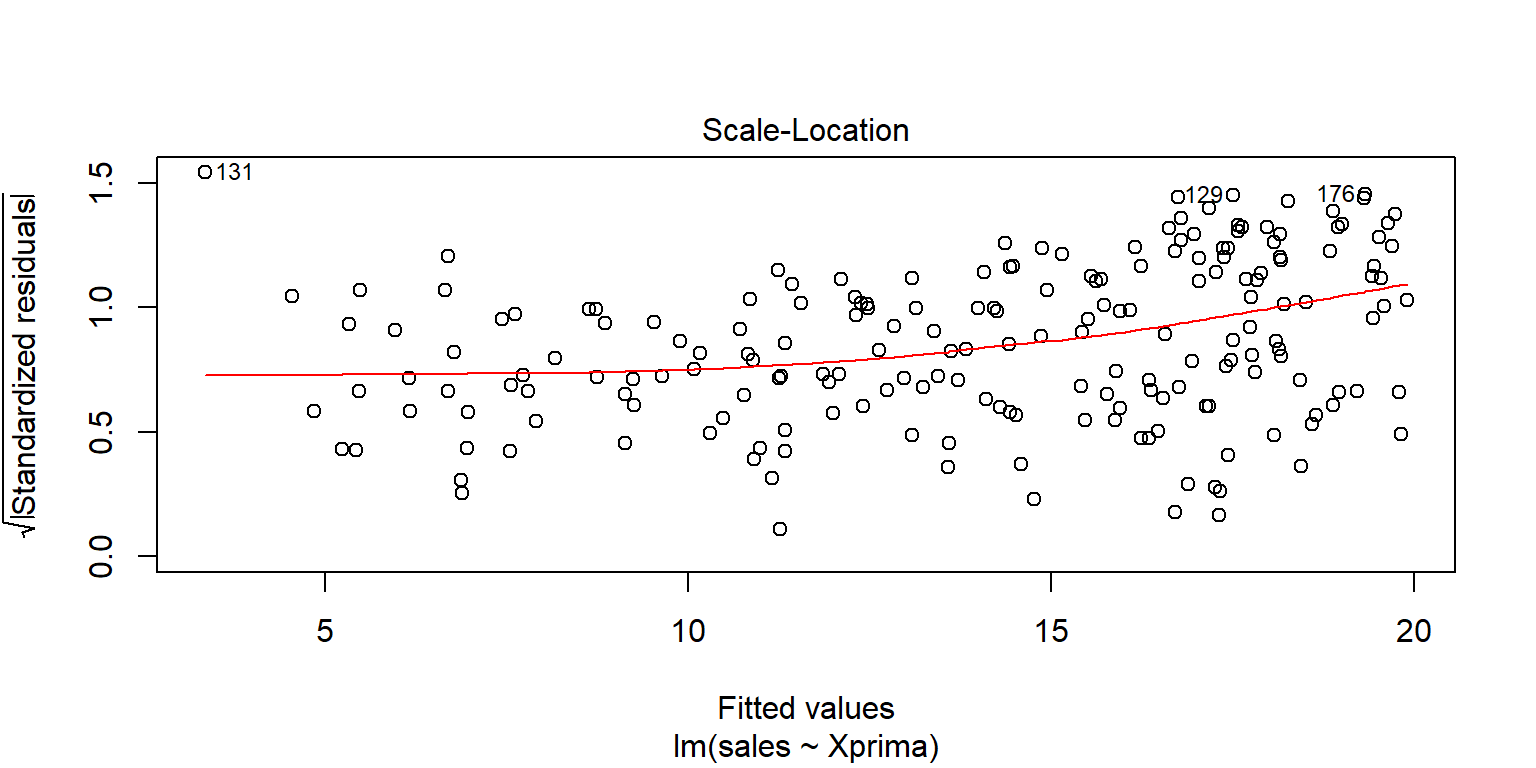
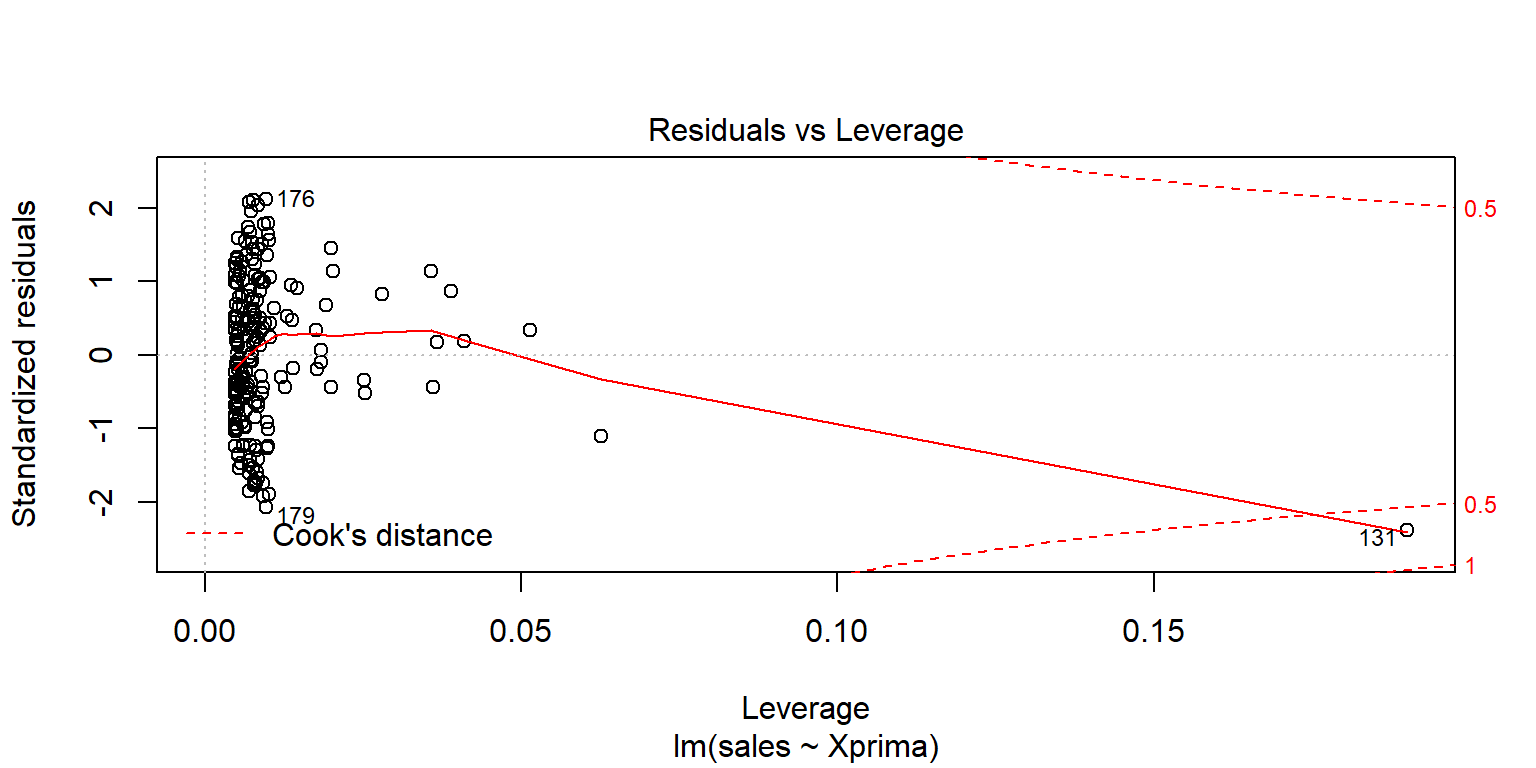
Datosfit2=augment(fit2pond)
head(Datosfit2)## # A tibble: 6 x 10
## sales Xprima X.weights. .fitted .se.fit .resid .hat .sigma .cooksd
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 22.1 15.2 0.0659 17.8 0.310 4.27 0.00798 0.890 6.14e-3
## 2 10.4 6.67 0.150 9.24 0.202 1.16 0.00770 0.893 9.90e-4
## 3 9.3 4.15 0.241 6.69 0.256 2.61 0.0199 0.889 2.13e-2
## 4 18.5 12.3 0.0812 14.9 0.232 3.56 0.00549 0.890 3.60e-3
## 5 12.9 13.4 0.0744 16.1 0.260 -3.19 0.00634 0.891 3.05e-3
## 6 7.2 2.95 0.339 5.48 0.289 1.72 0.0357 0.890 2.42e-2
## # ... with 1 more variable: .std.resid <dbl>normtest::jb.norm.test(Datosfit2$.std.resid)##
## Jarque-Bera test for normality
##
## data: Datosfit2$.std.resid
## JB = 4, p-value = 0.08nortest::lillie.test(Datosfit2$.std.resid)##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: Datosfit2$.std.resid
## D = 0.04, p-value = 0.5shapiro.test(Datosfit2$.std.resid)##
## Shapiro-Wilk normality test
##
## data: Datosfit2$.std.resid
## W = 1, p-value = 0.09fit2pond2 <- lm(sales~Xprima,weights = 1/(Xprima), data=Datos[-c(131),])
summary(fit2pond2)##
## Call:
## lm(formula = sales ~ Xprima, data = Datos[-c(131), ], weights = 1/(Xprima))
##
## Weighted Residuals:
## Min 1Q Median 3Q Max
## -1.7953 -0.6135 -0.0396 0.6488 1.9298
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 2.9427 0.4205 7.0 4e-11 ***
## Xprima 0.9727 0.0411 23.7 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.88 on 197 degrees of freedom
## Multiple R-squared: 0.74, Adjusted R-squared: 0.739
## F-statistic: 561 on 1 and 197 DF, p-value: <2e-16plot(fit2pond2)

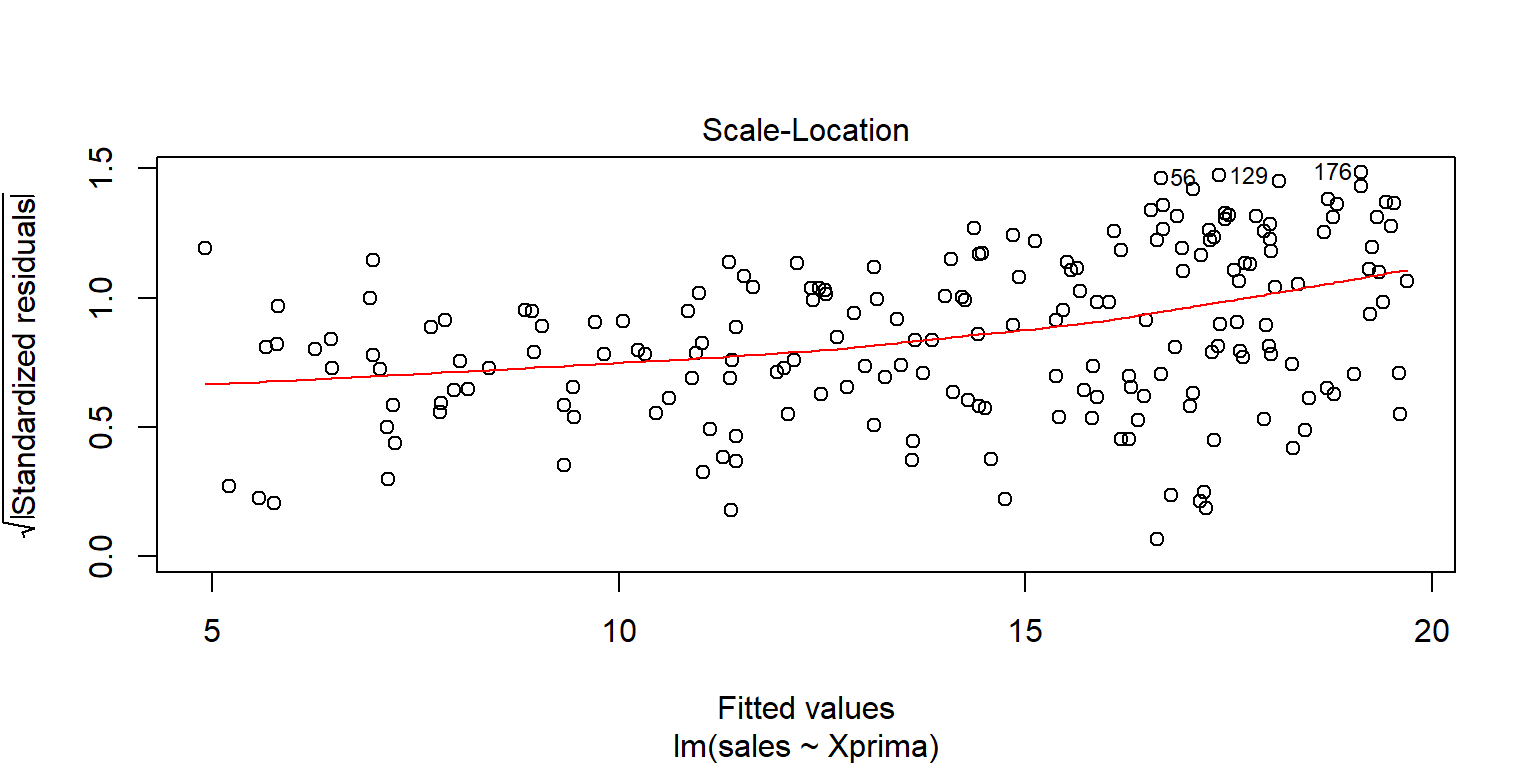
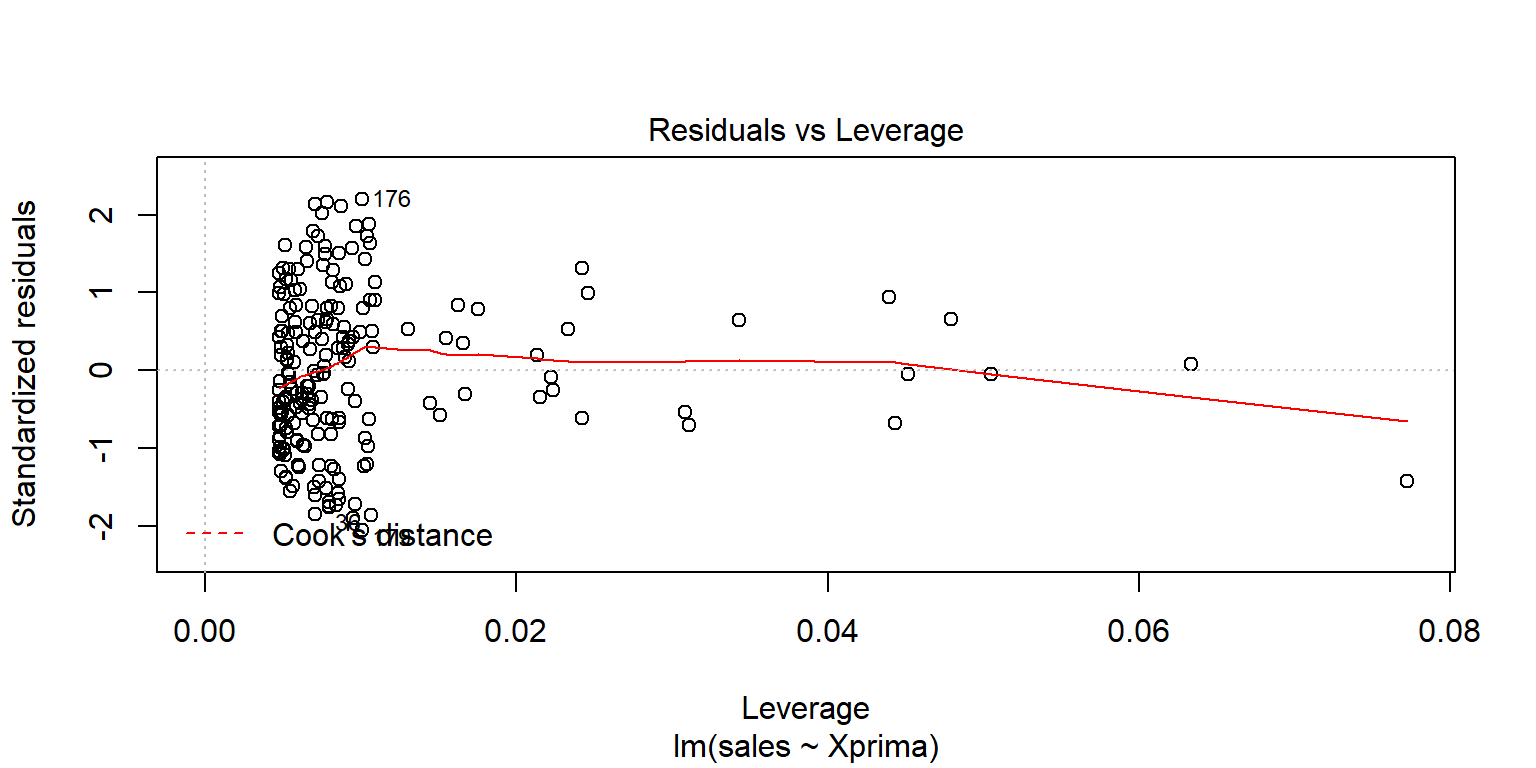
Datosfit2=augment(fit2pond2)
head(Datosfit2)## # A tibble: 6 x 11
## .rownames sales Xprima X.weights. .fitted .se.fit .resid .hat .sigma
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 22.1 15.2 0.0659 17.7 0.311 4.40 0.00823 0.879
## 2 2 10.4 6.67 0.150 9.43 0.214 0.969 0.00889 0.882
## 3 3 9.3 4.15 0.241 6.98 0.279 2.32 0.0242 0.879
## 4 4 18.5 12.3 0.0812 14.9 0.229 3.59 0.00550 0.880
## 5 5 12.9 13.4 0.0744 16.0 0.259 -3.12 0.00641 0.881
## 6 6 7.2 2.95 0.339 5.81 0.317 1.39 0.0439 0.881
## # ... with 2 more variables: .cooksd <dbl>, .std.resid <dbl>normtest::jb.norm.test(Datosfit2$.std.resid)##
## Jarque-Bera test for normality
##
## data: Datosfit2$.std.resid
## JB = 5, p-value = 0.06nortest::lillie.test(Datosfit2$.std.resid)##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: Datosfit2$.std.resid
## D = 0.05, p-value = 0.2shapiro.test(Datosfit2$.std.resid)##
## Shapiro-Wilk normality test
##
## data: Datosfit2$.std.resid
## W = 1, p-value = 0.03datonuevos= data.frame(Xprima=seq(0, 17, by=.2), sales=predict(fit2pond2, newdata=data.frame(Xprima=seq(0, 17, by=.2) ) ))
datonuevosold= data.frame(Xprima=seq(0, 17, by=.2), sales=predict(fit2pond, newdata=data.frame(Xprima=seq(0, 17, by=.2) ) ))
datonuevos$TV=(datonuevos$Xprima)^(2)
datonuevosold$TV=(datonuevos$Xprima)^(2)
ggplot(data=Datos, aes(x=TV,y=sales))+
geom_point(colour="black")+
geom_line(data= datonuevos, colour="green" )+
geom_line(data= datonuevosold, colour="red" )
ggplot(data=Datos, aes(x=Xprima,y=sales))+
geom_point(colour="black")+
geom_line(data= datonuevos, colour="green" )+
geom_line(data= datonuevosold, colour="red" )
Otra opción es tomar como pesos \(w=1/(6.33-1.96x+.18x^2)\)
fit2pondv2 <- lm(sales~Xprima,weights = 1/(6.33-1.96*Xprima+.18*Xprima^2), data=Datos)
summary(fit2pondv2)##
## Call:
## lm(formula = sales ~ Xprima, data = Datos, weights = 1/(6.33 -
## 1.96 * Xprima + 0.18 * Xprima^2))
##
## Weighted Residuals:
## Min 1Q Median 3Q Max
## -1.7604 -0.8913 -0.0963 0.7780 1.9610
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.3205 0.3376 9.83 <2e-16 ***
## Xprima 0.9345 0.0406 23.00 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1 on 198 degrees of freedom
## Multiple R-squared: 0.728, Adjusted R-squared: 0.726
## F-statistic: 529 on 1 and 198 DF, p-value: <2e-16plot(fit2pondv2)
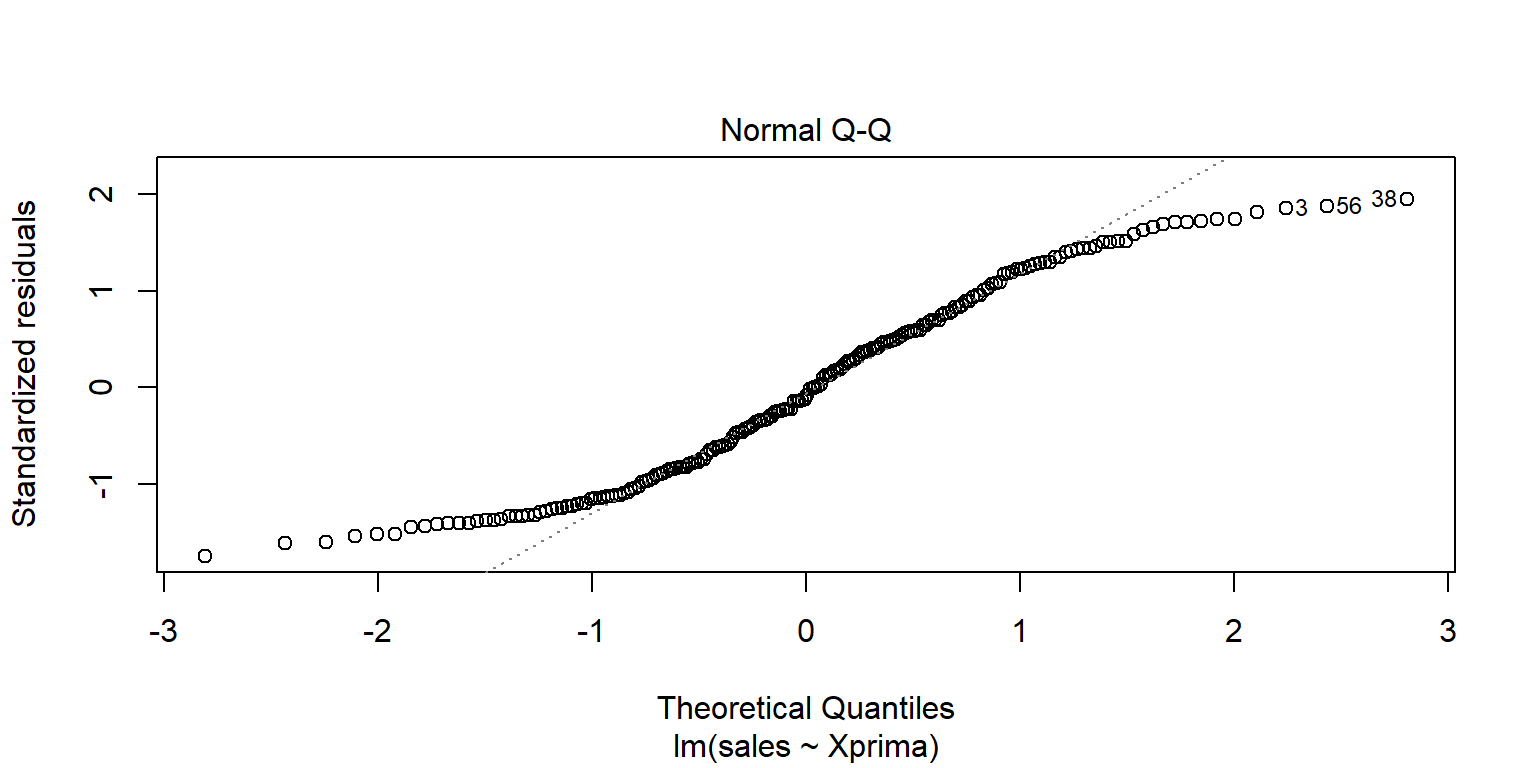
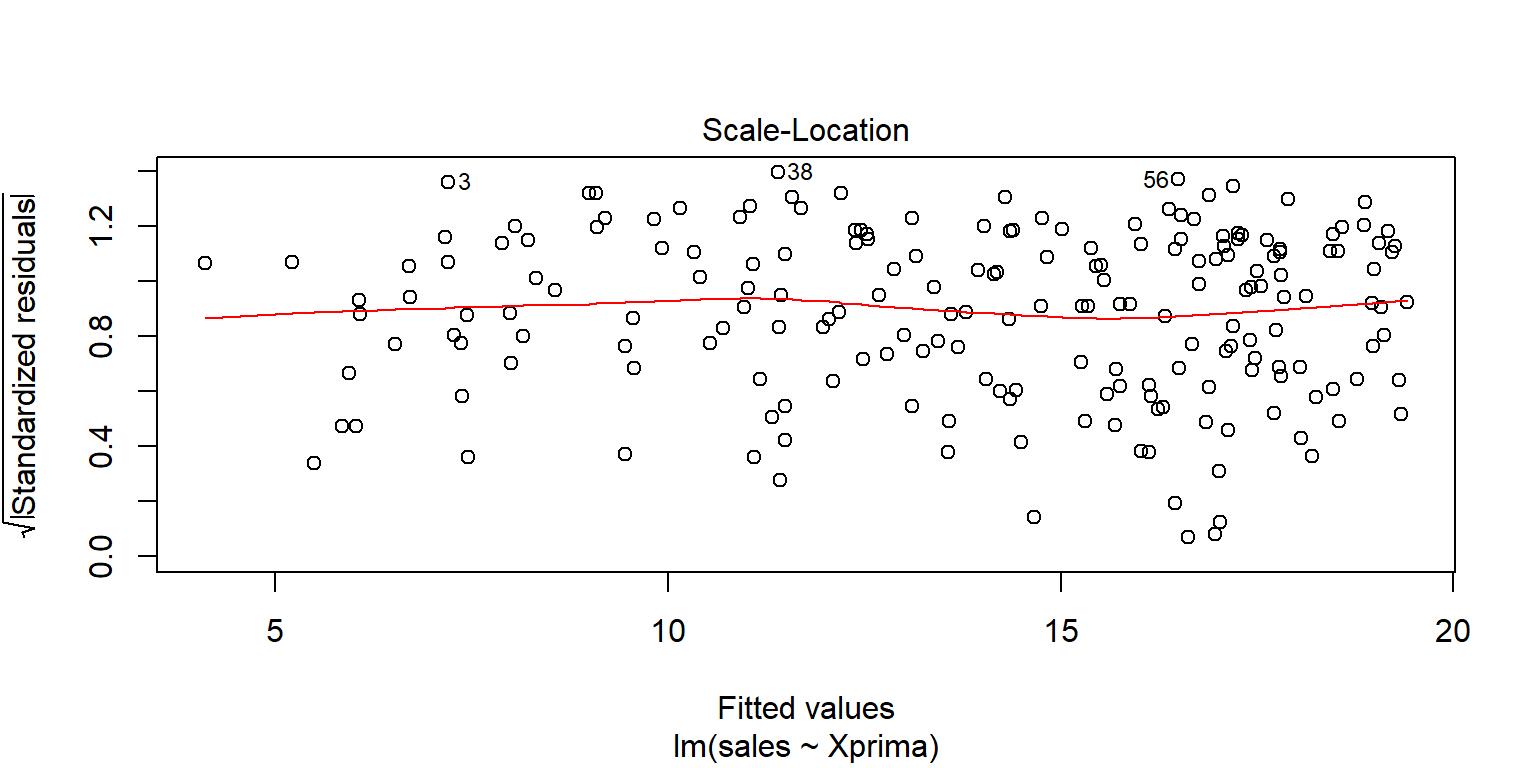

Datosfit2=augment(fit2pondv2)
head(Datosfit2)## # A tibble: 6 x 10
## sales Xprima X.weights. .fitted .se.fit .resid .hat .sigma .cooksd
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 22.1 15.2 0.0555 17.5 0.342 4.60 0.00635 1.01 0.00370
## 2 10.4 6.67 0.790 9.55 0.147 0.845 0.0168 1.01 0.00479
## 3 9.3 4.15 0.771 7.20 0.198 2.10 0.0297 1.00 0.0526
## 4 18.5 12.3 0.106 14.8 0.241 3.68 0.00601 1.01 0.00425
## 5 12.9 13.4 0.0799 15.9 0.280 -2.99 0.00612 1.01 0.00216
## 6 7.2 2.95 0.473 6.08 0.235 1.12 0.0255 1.01 0.00783
## # ... with 1 more variable: .std.resid <dbl>normtest::jb.norm.test(Datosfit2$.std.resid)##
## Jarque-Bera test for normality
##
## data: Datosfit2$.std.resid
## JB = 12, p-value = 0.008¿Quizás transformar Y?